Małe RNA tworzące spinki do włosów lub krótkie RNA tworzące spinki do włosów (shRNA krótkie RNA o strukturze spinki do włosów, małe RNA o strukturze spinki do włosów) cząsteczki krótkich RNA tworzące gęste spinki do włosów w strukturze drugorzędowej. ShRNA można wykorzystać do wyłączenia ekspresji... ... Wikipedia
Polimeraza RNA- z komórki T. aquaticus podczas replikacji. Niektóre elementy enzymu stają się przezroczyste, a łańcuchy RNA i DNA są wyraźniej widoczne. Jon magnezu (żółty) znajduje się w miejscu aktywnym enzymu. Polimeraza RNA to enzym, który wykonuje ... ... Wikipedia
Interferencja RNA- Dostarczanie małych RNA zawierających spinki do włosów za pomocą wektora na bazie lentiwirusa i mechanizm interferencji RNA w komórkach ssaków Interferencja RNA (a... Wikipedia
Gen RNA- Niekodujący RNA (ncRNA) to cząsteczki RNA, które nie ulegają translacji na białka. Poprzednio używany synonim, małe RNA (smRNA, małe RNA), nie jest już używany, ponieważ niektóre niekodujące RNA mogą być bardzo ... ... Wikipedia
Małe jądrowe RNA- (snRNA, snRNA) klasa RNA występująca w jądrze komórek eukariotycznych. Są transkrybowane przez polimerazę RNA II lub polimerazę RNA III i biorą udział w ważnych procesach, takich jak splicing (usuwanie intronów z niedojrzałego mRNA), regulacja… Wikipedia
Małe jąderkowe RNA- (snoRNA, ang. snoRNA) klasa małych RNA biorących udział w modyfikacjach chemicznych (metylacja i pseudourydylacja) rybosomalnego RNA, a także tRNA i małego jądrowego RNA. Według klasyfikacji MeSH małe jąderkowe RNA uznawane są za podgrupę... ...Wikipedia
małe jądrowe (jądrowe o niskiej masie cząsteczkowej) RNA- Rozległa grupa (105 106) małych jądrowych RNA (100 300 nukleotydów), związanych z heterogenicznym jądrowym RNA, jest częścią małych granulek rybonukleoproteinowych jądra; M.n.RNA są niezbędnym składnikiem systemu splicingu... ...
małe cytoplazmatyczne RNA- Małe (100-300 nukleotydów) cząsteczki RNA zlokalizowane w cytoplazmie, podobne do małego jądrowego RNA. [Arefyev V.A., Lisovenko L.A. Angielsko-rosyjski słownik objaśniający terminy genetyczne 1995 407 s.] Tematy genetyka EN scyrpssmall cytoplasmic... ... Przewodnik tłumacza technicznego
małe jądrowe RNA klasy U- Grupa związanych z białkami małych (od 60 do 400 nukleotydów) cząsteczek RNA, które stanowią znaczną część zawartości spplikomu i biorą udział w procesie wycinania intronów; w 4 z 5 dobrze zbadanych typów Usn, RNA U1, U2, U4 i U5 to 5... ... Przewodnik tłumacza technicznego
Biomarkery RNA- * Biomarkery RNA * Biomarkery RNA to ogromna liczba ludzkich transkryptów, które nie kodują syntezy białek (nsbRNA lub npcRNA). W większości przypadków małe (miRNA, snoRNA) i długie (antysensowne RNA, dsRNA i inne typy) cząsteczki RNA to... ... Genetyka. słownik encyklopedyczny
Książki
- Kup za 1877 UAH (tylko Ukraina)
- Genetyka kliniczna. Podręcznik (+CD), Bochkov Nikolay Pavlovich, Puzyrev Valery Pavlovich, Smirnikhina Svetlana Anatolyevna. Wszystkie rozdziały zostały poprawione i uzupełnione w związku z rozwojem nauk i praktyki medycznej. Rozdziały poświęcone chorobom wieloczynnikowym, profilaktyce, leczeniu chorób dziedzicznych,…
), zapobiegając translacji mRNA na rybosomach na kodowane przez nie białko. Ostatecznie efekt małego interferującego RNA jest identyczny jak zwykłe zmniejszenie ekspresji genów.
Małe interferujące RNA zostały odkryte w 1999 roku przez grupę Davida Baulcombe w Wielkiej Brytanii jako składnik potranskrypcyjnego systemu wyciszania genów u roślin. PTGS, en: posttranskrypcyjne wyciszanie genów). Zespół opublikował swoje odkrycia w czasopiśmie Science.
Dwuniciowy RNA może zwiększać ekspresję genów poprzez mechanizm zwany aktywacją genów zależną od RNA. RNAa, aktywacja genów indukowana małym RNA). Wykazano, że dwuniciowe RNA komplementarne do promotorów genów docelowych powodują aktywację odpowiednich genów. W przypadku komórek ludzkich wykazano aktywację zależną od RNA po podaniu syntetycznego dwuniciowego RNA. Nie wiadomo, czy podobny system istnieje w komórkach innych organizmów.
Zapewniając możliwość wyłączenia zasadniczo dowolnego genu według własnego uznania, mała interferująca interferencja RNA oparta na RNA wywołała ogromne zainteresowanie biologią podstawową i stosowaną. Rośnie liczba szeroko zakrojonych testów opartych na RNAi, służących do identyfikacji ważnych genów w szlakach biochemicznych. Ponieważ o rozwoju chorób determinuje także aktywność genów, oczekuje się, że w niektórych przypadkach wyłączenie genu za pomocą małego interferującego RNA może mieć efekt terapeutyczny.
Jednakże zastosowanie interferencji RNA opartej na małym interferującym RNA u zwierząt, a zwłaszcza u ludzi, napotyka wiele trudności. Eksperymenty wykazały, że skuteczność małych interferujących RNA jest różna różne rodzaje komórki: niektóre komórki łatwo reagują na mały interferujący RNA i wykazują spadek ekspresji genów, podczas gdy w innych nie obserwuje się tego pomimo skutecznej transfekcji. Przyczyny tego zjawiska są nadal słabo poznane.
Wyniki badań I fazy pierwszych dwóch leków RNAi (przeznaczonych do leczenia zwyrodnienia plamki żółtej), opublikowane pod koniec 2005 roku, pokazują, że leki zawierające małe interferujące RNA są łatwo tolerowane przez pacjentów i mają akceptowalne właściwości farmakokinetyczne.
Wstępne badania kliniczne małych interferujących RNA ukierunkowanych na wirusa Ebola wskazują, że mogą one być skuteczne w profilaktyce choroby po ekspozycji. Lek ten pozwolił całej grupie eksperymentalnych naczelnych przeżyć po otrzymaniu śmiertelnej dawki wirusa Ebola Zairu
Metafora leżąca u podstaw nazwy zjawiska interferencji RNA nawiązuje do eksperymentu z petunią, kiedy sztucznie wprowadzone do rośliny geny syntetazy pigmentu różowego i fioletowego nie zwiększały intensywności barwy, a wręcz przeciwnie, ją zmniejszały. Podobnie w przypadku „zwykłej” interferencji nałożenie dwóch fal może prowadzić do wzajemnego „zniesienia”.
W żywej komórce przepływ informacji między jądrem a cytoplazmą nigdy nie wysycha, jednak zrozumienie wszystkich jej „zawirowań” i rozszyfrowanie zakodowanej w niej informacji to naprawdę herkulesowe zadanie. Za jeden z najważniejszych przełomów w biologii ostatniego stulecia można uznać odkrycie informacyjnych (lub matrycowych) cząsteczek RNA (mRNA lub mRNA), które służą jako pośrednicy przenoszący „wiadomości” informacyjne z jądra (z chromosomów) do cytoplazmy . Decydującą rolę RNA w syntezie białek przepowiedziano już w 1939 r. w pracach Torbjörna Casperssona, Jeana Bracheta i Jacka Schultza, a w 1971 r. George Marbaix zapoczątkował syntezę hemoglobiny w oocytach żab poprzez wstrzyknięcie pierwszego wyizolowanego króliczego informacyjnego RNA kodującego to białko .
W latach 1956-57 w Związku Radzieckim A. N. Belozersky i A. S. Spirin niezależnie udowodnili istnienie mRNA, a także odkryli, że większość RNA w komórce to nie matryca, ale rybosomalny RNA (rRNA). Rybosomalny RNA, drugi „główny” typ RNA komórkowego, tworzy „szkielet” i centrum funkcjonalne rybosomów we wszystkich organizmach; To rRNA (a nie białka) reguluje główne etapy syntezy białek. Jednocześnie opisano i zbadano trzeci „główny” typ RNA - transferowe RNA (tRNA), które w połączeniu z dwoma innymi - mRNA i rRNA - tworzą pojedynczy kompleks syntetyzujący białko. Według dość popularnej hipotezy „świata RNA”, to właśnie ten kwas nukleinowy leżał u samych początków życia na Ziemi.
Dzięki temu, że RNA jest znacznie bardziej hydrofilowe w porównaniu do DNA (w wyniku zastąpienia dezoksyrybozy rybozą), jest bardziej labilne i może stosunkowo swobodnie przemieszczać się w komórce, a co za tym idzie dostarczać repliki krótkotrwałe Informacja genetyczna(mRNA) do miejsca, w którym rozpoczyna się synteza białka. Warto jednak zwrócić uwagę na związaną z tym „niedogodność” – RNA jest bardzo niestabilne. Jest przechowywany znacznie gorzej niż DNA (nawet wewnątrz komórki) i ulega degradacji przy najmniejszej zmianie warunków (temperatura, pH). Oprócz „własnej” niestabilności, ogromny wkład należy do rybonukleaz (lub RNaz) – klasy enzymów rozszczepiających RNA, które są bardzo stabilne i „wszechobecne” – nawet skóra dłoni eksperymentatora zawiera wystarczającą ilość tych enzymów, aby zanegować cały eksperyment. Z tego powodu praca z RNA jest znacznie trudniejsza niż z białkami czy DNA – to ostatnie można na ogół przechowywać przez setki tysięcy lat praktycznie bez uszkodzeń.
Fantastyczna opieka podczas pracy, tridestylat, sterylne rękawiczki, jednorazowe szkło laboratoryjne – to wszystko było niezbędne, aby zapobiec degradacji RNA, jednak utrzymanie takich standardów nie zawsze było możliwe. Dlatego przez długi czas po prostu nie zwracali uwagi na krótkie „fragmenty” RNA, które nieuchronnie zanieczyszczały roztwory. Jednak z biegiem czasu stało się jasne, że pomimo wszelkich wysiłków zmierzających do utrzymania sterylności miejsca pracy, w naturalny sposób nadal odkrywane są „zanieczyszczenia”, a potem okazało się, że w cytoplazmie zawsze znajdują się tysiące krótkich dwuniciowych RNA , pełniąc bardzo specyficzne funkcje i są absolutnie niezbędne do prawidłowego rozwoju komórek i organizmu.
Zasada interferencji RNA
Obecnie badanie małych regulatorowych RNA jest jednym z najszybciej rozwijających się obszarów Biologia molekularna. Odkryto, że wszystkie krótkie RNA spełniają swoje funkcje w oparciu o zjawisko zwane interferencją RNA (istotą tego zjawiska jest tłumienie ekspresji genów na etapie transkrypcji lub translacji przy aktywnym udziale małych cząsteczek RNA). Mechanizm interferencji RNA pokazano bardzo schematycznie na ryc. 1:
Ryż. 1. Podstawy interferencji RNA
Cząsteczki dwuniciowego RNA (dsRNA) są rzadkością w normalnych komórkach, ale stanowią istotny etap w cyklu życiowym wielu wirusów. Specjalne białko zwane Dicer, po wykryciu dsRNA w komórce, „tnie” je na małe fragmenty. Nić antysensowna takiego fragmentu, który można już nazwać krótkim interferującym RNA (siRNA, od siRNA – small interferencyjny RNA), jest związana przez kompleks białek zwany RISC (ang. RNA-operative Silencing Complex), którego centralnym elementem jest endonukleaza z rodziny Argonaute. Wiązanie z siRNA aktywuje RISC i inicjuje poszukiwanie w komórce cząsteczek DNA i RNA, które są komplementarne do „wzorcowego” siRNA. Losem takich cząsteczek jest zniszczenie lub inaktywacja przez kompleks RISC.
Podsumowując, krótkie „cięcia” obcego (w tym celowo wprowadzonego) dwuniciowego RNA służą jako „szablon” do poszukiwania na dużą skalę i niszczenia komplementarnego mRNA (co jest równoznaczne z tłumieniem ekspresji odpowiedniego genu) nie tylko w jednej komórce, ale także w sąsiednich. Dla wielu organizmów - pierwotniaków, mięczaków, robaków, owadów, roślin - zjawisko to jest jednym z głównych sposobów obrony immunologicznej przed infekcjami.
W 2006 roku Andrew Fire i Craig Mello otrzymali nagroda Nobla Doktorat z Fizjologii i Medycyny „Za odkrycie zjawiska interferencji RNA – mechanizmu wyciszania genów przy udziale dsRNA”. Choć samo zjawisko interferencji RNA opisano już dużo wcześniej (na początku lat 80. XX w.), to dopiero za sprawą Fire’a i Mello w Ogólny zarys określił mechanizm regulacyjny małych RNA i nakreślił nieznany dotąd obszar badań molekularnych. Oto główne rezultaty ich pracy:
- Podczas interferencji RNA rozszczepiane jest mRNA (a nie inne);
- Dwuniciowy RNA działa (powoduje rozszczepienie) znacznie skuteczniej niż jednoniciowy RNA. Te dwie obserwacje przewidywały istnienie wyspecjalizowanego układu pośredniczącego w działaniu dsRNA;
- dsRNA, komplementarny do odcinka dojrzałego mRNA, powoduje rozszczepienie tego ostatniego. Wskazywało to na cytoplazmatyczną lokalizację procesu i obecność specyficznej endonukleazy;
- Niewielka ilość dsRNA (kilka cząsteczek na komórkę) wystarczy, aby całkowicie „wyłączyć” gen docelowy, co wskazuje na istnienie kaskadowego mechanizmu katalizy i/lub amplifikacji.
Wyniki te położyły podwaliny pod cały obszar współczesnej biologii molekularnej – interferencję RNA – i na dziesięciolecia wyznaczyły wektor prac wielu grup badawczych na całym świecie. Do chwili obecnej odkryto trzy duże grupy małych RNA, które odgrywają w polu molekularnym rolę „zespołu interferującego RNA”. Poznajmy je bardziej szczegółowo.
Gracz nr 1 – krótki interferujący RNA
O specyfice interferencji RNA decyduje krótki interferujący RNA (siRNA) – małe dwuniciowe cząsteczki RNA o wyraźnie określonej strukturze (patrz ryc. 2).
siRNA są najwcześniejsze w ewolucji i są najbardziej rozpowszechnione w roślinach, organizmach jednokomórkowych i bezkręgowcach. U kręgowców praktycznie nie stwierdza się siRNA, ponieważ zostały one zastąpione późniejszymi „modelami” krótkich RNA (patrz poniżej).
siRNA – „szablony” do przeszukiwania cytoplazmy i niszczenia cząsteczek mRNA – mają długość 20–25 nukleotydów i „cechę szczególną”: 2 niesparowane nukleotydy na końcach 3’ i fosforylowane końce 5’. Antysensowny siRNA jest w stanie (oczywiście nie sam, ale przy pomocy kompleksu RISC) rozpoznać mRNA i specyficznie spowodować jego degradację: docelowy mRNA jest cięty dokładnie w miejscu komplementarnym do 10. i 11. nukleotydu antysensowny łańcuch siRNA.

Ryż. 2. Mechanizm „interferencji” pomiędzy mRNA i siRNA
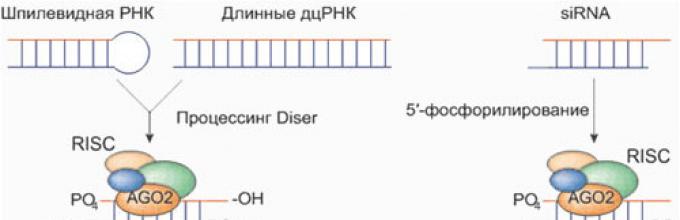
„Interferujące” krótkie cząsteczki RNA mogą albo przedostać się do komórki z zewnątrz, albo zostać „wycięte” z dłuższego dwuniciowego RNA. Głównym białkiem wymaganym do cięcia dsRNA jest endonukleaza Dicer. „Wyłączenie” genu poprzez mechanizm interferencyjny odbywa się za pomocą siRNA wraz z kompleksem białkowym RISC, na który składają się trzy białka – endonukleaza Ago2 oraz dwa białka pomocnicze PACT i TRBP. Później odkryto, że kompleksy Dicer i RISC mogą wykorzystywać jako „nasiono” nie tylko dsRNA, ale także jednoniciowy RNA tworzący dwuniciową spinkę do włosów, a także gotowy siRNA (ten ostatni omija „cięcie” etapie i natychmiast wiąże się z RISC).
Funkcje siRNA w komórkach bezkręgowców są dość zróżnicowane. Pierwszą i najważniejszą rzeczą jest ochrona immunologiczna. „Tradycyjny” układ odpornościowy (limfocyty + leukocyty + makrofagi) występuje tylko w złożonych organizmach wielokomórkowych. U organizmów jednokomórkowych, bezkręgowców i roślin (które albo nie mają takiego systemu, albo jest on w powijakach) obrona immunologiczna opiera się na interferencji RNA. Odporność oparta na interferencji RNA nie wymaga skomplikowanych narządów „trenujących” prekursorów komórek odpornościowych (śledziona, grasica); jednocześnie różnorodność teoretycznie możliwych krótkich sekwencji RNA (421 wariantów) jest skorelowana z liczbą możliwych przeciwciał białkowych zwierząt wyższych. Ponadto siRNA syntetyzowane są na bazie „wrogiego” RNA, który zainfekował komórkę, co oznacza, że w przeciwieństwie do przeciwciał są natychmiast „dopasowywane” do konkretnego rodzaju infekcji. I choć ochrona oparta na interferencji RNA nie działa poza komórką (przynajmniej nie ma jeszcze takich danych), to zapewnia odporność wewnątrzkomórkową bardziej niż zadowalająco.
Przede wszystkim siRNA tworzy odporność przeciwwirusową poprzez niszczenie mRNA lub genomowego RNA organizmów zakaźnych (tak np. odkryto siRNA w roślinach). Wprowadzenie wirusowego RNA powoduje silną amplifikację specyficznych siRNA w oparciu o cząsteczkę startera - sam wirusowy RNA. Ponadto siRNA tłumią ekspresję różnych ruchomych elementów genetycznych (MGE), a zatem zapewniają ochronę przed endogennymi „infekcjami”. Mutacje w genach kompleksu RISC często prowadzą do zwiększonej niestabilności genomu ze względu na wysoką aktywność MGE; siRNA może działać jako ogranicznik ekspresji własnych genów, uruchamiając się w odpowiedzi na ich nadekspresję. Regulacja funkcji genów może zachodzić nie tylko na poziomie translacji, ale także podczas transkrypcji – poprzez metylację genów na histonie H3.
We współczesnej biologii eksperymentalnej nie można przecenić znaczenia interferencji RNA i krótkich RNA. Opracowano technologię „wyłączania” (lub niszczenia) poszczególnych genów in vitro (na hodowlach komórkowych) i in vivo (na embrionach), co stało się już de facto standardem przy badaniu dowolnego genu. Czasem nawet w celu ustalenia roli poszczególnych genów w jakimś procesie systematycznie „wyłączają” wszystkie geny po kolei.
Możliwość wykorzystania siRNA zainteresowali się także farmaceuci, gdyż zdolność do specyficznej regulacji funkcjonowania poszczególnych genów stwarza niespotykane dotąd perspektywy w leczeniu wielu chorób. Mały rozmiar i duża specyficzność działania zapewniają wysoką skuteczność i niską toksyczność leków opartych na siRNA; Jednak nie udało się jeszcze rozwiązać problemu dostarczania siRNA do chorych komórek w organizmie - wynika to z kruchości i kruchości tych cząsteczek. I chociaż dziesiątki zespołów próbują obecnie znaleźć sposób na skierowanie tych „magicznych kul” dokładnie w cel (wewnątrz chorych narządów), nie osiągnęły one jeszcze widocznego sukcesu. Poza tym są inne trudności. Przykładowo w przypadku terapii przeciwwirusowej wysoka selektywność działania siRNA może wyrządzić krzywdę – skoro wirusy szybko mutują, zmodyfikowany szczep bardzo szybko straci wrażliwość na wybrany na początku terapii siRNA: wiadomo, że zastąpienie tylko jednego nukleotydu w siRNA prowadzi do znacznego zmniejszenia efektu interferencji.
W tym miejscu warto jeszcze raz przypomnieć – siRNA znaleziono jedynie u roślin, bezkręgowców i organizmów jednokomórkowych; Chociaż homologi białek do interferencji RNA (Dicer, kompleks RISC) są również obecne u zwierząt wyższych, siRNA nie zostały wykryte konwencjonalnymi metodami. Cóż za niespodzianka, gdy sztucznie wprowadzone syntetyczne analogi siRNA wywołały silny, specyficzny, zależny od dawki efekt w hodowlach komórkowych ssaków! Oznaczało to, że w komórkach kręgowców interferencja RNA nie została zastąpiona przez bardziej złożony układ odpornościowy, ale ewoluowała wraz z organizmami, przekształcając się w coś bardziej „zaawansowanego”. W związku z tym u ssaków konieczne było poszukiwanie nie dokładnych analogów siRNA, ale ich ewolucyjnych następców.
Gracz nr 2 – mikroRNA
Rzeczywiście, w oparciu o starożytny ewolucyjnie mechanizm interferencji RNA, bardziej rozwinięte organizmy rozwinęły dwa wyspecjalizowane systemy kontrolowania działania genów, każdy wykorzystujący własną grupę małych RNA - mikroRNA i piRNA (RNA oddziałujący z Piwi). Obydwa systemy pojawiły się w gąbkach i koelenteratach i wraz z nimi ewoluowały, wypierając siRNA i mechanizm interferencji „nagiego” RNA. Ich rola w zapewnianiu odporności maleje, gdyż funkcję tę przejmują bardziej zaawansowane mechanizmy odporności komórkowej, w szczególności układ interferonowy. Jednakże system ten jest na tyle czuły, że uruchamia także sam siRNA: pojawienie się małego dwuniciowego RNA w komórce ssaka wyzwala „sygnał alarmowy” (aktywuje wydzielanie interferonu i powoduje ekspresję genów zależnych od interferonu, co całkowicie blokuje wszystkie procesy tłumaczeniowe). Pod tym względem w mechanizmie interferencji RNA u zwierząt wyższych pośredniczą głównie mikroRNA i piRNA - jednoniciowe cząsteczki o specyficznej strukturze, które nie są wykrywane przez układ interferonu.
W miarę jak genom stawał się coraz bardziej złożony, mikroRNA i piRNA w coraz większym stopniu angażowały się w regulację transkrypcji i translacji. Z biegiem czasu zamieniły się w dodatkowy, precyzyjny i subtelny system regulacji genomu. W przeciwieństwie do siRNA, mikroRNA i piRNA (odkryte w 2001 r., patrz ryc. 3, A-B) nie są wytwarzane z obcych cząsteczek dwuniciowego RNA, ale są początkowo kodowane w genomie organizmu gospodarza.
Prekursor mikroRNA ulega transkrypcji z obu nici genomowego DNA przez polimerazę RNA II, co skutkuje pojawieniem się formy pośredniej – pri-microRNA – noszącej cechy zwykłego mRNA – czapeczki m7G i ogona poliA. Prekursor ten tworzy pętlę z dwoma jednoniciowymi „ogonami” i kilkoma niesparowanymi nukleotydami w środku (ryc. 3A). Taka pętla poddawana jest dwuetapowej obróbce (ryc. B): najpierw endonukleaza Drosha odcina jednoniciowe „ogony” RNA od spinki do włosów, po czym wycięty spinka do włosów (pre-microRNA) jest eksportowany do cytoplazmy, gdzie jest rozpoznawany przez Dicera, który wykonuje jeszcze dwa cięcia (wycinany jest odcinek dwuniciowy, oznaczony kolorami na ryc. 3A). W tej postaci dojrzały mikroRNA, podobnie jak siRNA, wchodzi w skład kompleksu RISC.
Mechanizm działania wielu mikroRNA jest podobny do działania siRNA: krótki (21–25 nukleotydów) jednoniciowy RNA będący częścią kompleksu białkowego RISC wiąże się z dużą swoistością z miejscem komplementarnym w nieulegającym translacji regionie 3' docelowy mRNA. Wiązanie prowadzi do rozszczepienia mRNA przez białko Ago. Jednak aktywność mikroRNA (w porównaniu z siRNA) jest już bardziej zróżnicowana – jeśli komplementarność nie jest absolutna, docelowy mRNA może nie ulec degradacji, a jedynie odwracalnie zablokowany (nie będzie translacji). Ten sam kompleks RISC może również wykorzystywać sztucznie wprowadzone siRNA. To wyjaśnia, dlaczego siRNA utworzone przez analogię z pierwotniakami są również aktywne u ssaków.
W ten sposób możemy uzupełnić ilustrację mechanizmu działania interferencji RNA w organizmach wyższych (dwustronnie symetrycznych), łącząc na jednym rysunku diagram działania mikroRNA i siRNA wprowadzonych biotechnologicznie (ryc. 3B).
Ryż. 3A: Struktura dwuniciowej cząsteczki prekursora mikroRNA
Główne cechy: obecność konserwatywnych sekwencji tworzących spinkę do włosów; obecność kopii komplementarnej (microRNA*) z dwoma „dodatkowymi” nukleotydami na końcu 3’; specyficzna sekwencja (2–8 pz), która tworzy miejsce rozpoznawane przez endonukleazy. Sam mikroRNA jest podświetlony na czerwono — to właśnie wycina Dicer.

Ryż. Ryc. 3B: Ogólny mechanizm przetwarzania mikroRNA i realizacja jego działania

Ryż. 3B: Uogólniony schemat działania sztucznych mikroRNA i siRNA
Sztuczne siRNA wprowadza się do komórki za pomocą wyspecjalizowanych plazmidów (wektor ukierunkowany na siRNA).
Funkcje mikroRNA
Funkcje fizjologiczne mikroRNA są niezwykle zróżnicowane - w rzeczywistości pełnią one rolę głównych niebiałkowych regulatorów ontogenezy. mikroRNA nie znoszą, ale uzupełniają „klasyczny” schemat regulacji genów (induktory, supresory, zagęszczenie chromatyny itp.). Ponadto synteza samych mikroRNA jest kompleksowo regulowana (niektóre pule mikroRNA mogą być włączane przez interferony, interleukiny, czynnik martwicy nowotworu α (TNF-α) i wiele innych cytokin). W rezultacie powstaje wielopoziomowa sieć strojenia „orkiestry” tysięcy genów, zadziwiającej swoją złożonością i elastycznością, ale na tym się nie kończy.
mikroRNA są bardziej „uniwersalne” niż siRNA: geny „oddziałowe” nie muszą być w 100% komplementarne – regulacja odbywa się również poprzez częściową interakcję. Obecnie jednym z najgorętszych tematów w biologii molekularnej jest poszukiwanie mikroRNA, które pełnią rolę alternatywnych regulatorów znanych procesów fizjologicznych. Na przykład opisano już mikroRNA zaangażowane w regulację cyklu komórkowego i apoptozę u roślin, Drosophila i nicieni; u ludzi mikroRNA regulują układ odpornościowy i rozwój krwiotwórczych komórek macierzystych. Zastosowanie technologii opartych na biochipach (przesiewowe mikromacierze) pokazało, że całe pule małych RNA są włączane i wyłączane na różnych etapach życia komórki. Dla procesy biologiczne zidentyfikowali dziesiątki specyficznych mikroRNA, których poziom ekspresji w określonych warunkach zmienia się tysiące razy, podkreślając wyjątkową sterowalność tych procesów.
Do niedawna uważano, że mikroRNA jedynie tłumią – całkowicie lub częściowo – pracę genów. Niedawno okazało się jednak, że działanie mikroRNA może się radykalnie różnić w zależności od stanu komórki! W aktywnie dzielącej się komórce mikroRNA wiąże się z komplementarną sekwencją w regionie 3' mRNA i hamuje syntezę białek (translację). Jednak w stanie spoczynku lub stresu (na przykład podczas uprawy w złym środowisku) to samo zdarzenie prowadzi do dokładnie odwrotnego efektu - zwiększonej syntezy docelowego białka!
Ewolucja mikroRNA
Liczba odmian mikroRNA w organizmach wyższych nie została jeszcze w pełni ustalona, według niektórych danych przekracza 1% liczby genów kodujących białka (u człowieka np. mówią, że jest ich 700 i jest to liczba stale rośnie). mikroRNA regulują aktywność około 30% wszystkich genów (dla wielu z nich cele nie są jeszcze znane), a istnieją cząsteczki zarówno wszechobecne, jak i specyficzne tkankowo - na przykład jedna z takich ważnych puli mikroRNA reguluje dojrzewanie pnia krwi komórki.
Szeroki profil ekspresji w różnych tkankach różne organizmy a biologiczna obfitość mikroRNA sugeruje starożytne ewolucyjne pochodzenie. MikroRNA odkryto po raz pierwszy u nicieni i przez długi czas uważano, że cząsteczki te występują jedynie w gąbkach i koelenteratach; jednakże odkryto je później w algach jednokomórkowych. Co ciekawe, w miarę jak organizmy stają się coraz bardziej złożone, wzrasta również liczba i niejednorodność puli miRNA. Pośrednio wskazuje to, że złożoność tych organizmów zapewnia w szczególności funkcjonowanie mikroRNA. Możliwą ewolucję miRNA pokazano na ryc. 4.

Ryż. 4. Różnorodność mikroRNA w różnych organizmach
Im wyższa organizacja organizmu, tym więcej znajduje się w nim mikroRNA (liczba w nawiasach). Gatunki, u których znaleziono pojedyncze mikroRNA, zaznaczono na czerwono. Według .
Można wyciągnąć wyraźne powiązanie ewolucyjne pomiędzy siRNA i mikroRNA w oparciu o następujące fakty:
- działanie obu typów jest wymienne i pośredniczą w nim białka homologiczne;
- siRNA wprowadzone do komórek ssaków specyficznie „wyłączają” pożądane geny (pomimo pewnej aktywacji ochrony interferonem);
- MikroRNA odkrywa się w coraz starszych organizmach.
Te i inne dane sugerują pochodzenie obu systemów od wspólnego „przodka”. Warto również zauważyć, że odporność „RNA” jako niezależny prekursor przeciwciał białkowych potwierdza teorię pochodzenia pierwszych form życia opartą na RNA, a nie na białkach (przypomnijmy, że jest to ulubiona teoria akademika A.S. Spirina) .
Chociaż na arenie biologii molekularnej było tylko dwóch „graczy” – siRNA i mikroRNA – główny „cel” interferencji RNA wydawał się całkowicie jasny. Rzeczywiście: zestaw homologicznych krótkich RNA i białek w różnych organizmach wykonuje podobne działania; W miarę jak organizmy stają się coraz bardziej złożone, wzrasta także funkcjonalność.
Jednak w procesie ewolucji natura stworzyła inny, ewolucyjnie najnowszy i wysoce wyspecjalizowany system oparty na tej samej skutecznej zasadzie interferencji RNA. Mówimy o piRNA (piRNA, z RNA interakcji Piwi).
Im bardziej złożony jest genom, tym bardziej rozwinięty i przystosowany jest organizm (lub odwrotnie? ;-). Jednak rosnąca złożoność genomu ma również wadę: złożony system genetyczny staje się niestabilny. Prowadzi to do konieczności istnienia mechanizmów odpowiedzialnych za utrzymanie integralności genomu – w przeciwnym razie spontaniczne „mieszanie” DNA po prostu go unieruchomi. Ruchome elementy genetyczne (MGE), jeden z głównych czynników niestabilności genomu, to krótkie, niestabilne regiony, które mogą ulegać autonomicznej transkrypcji i migrować w całym genomie. Aktywacja takich elementów transpozycyjnych prowadzi do wielokrotnych pęknięć DNA w chromosomach, co może mieć śmiertelne konsekwencje.
Liczba MGE rośnie nieliniowo wraz z rozmiarem genomu, a ich aktywność musi być ograniczona. Aby to zrobić, zwierzęta, zaczynając od koelenteratów, wykorzystują to samo zjawisko interferencji RNA. Tę funkcję pełnią także krótkie RNA, ale nie te już omówione, a trzeci typ – piRNA.
„Portret” piRNA
piRNA to krótkie cząsteczki o długości 24–30 nukleotydów, kodowane w regionach centromerowych i telomerowych chromosomu. Sekwencje wielu z nich są komplementarne do znanych ruchomych elementów genetycznych, ale istnieje wiele innych piRNA, które pokrywają się z regionami działających genów lub z fragmentami genomu, których funkcje są nieznane.
piRNA (a także mikroRNA) są kodowane w obu niciach genomowego DNA; są bardzo zmienne i różnorodne (nawet 500 000 (!) gatunków w jednym organizmie). W przeciwieństwie do siRNA i mikroRNA, są one utworzone przez jeden łańcuch cecha charakterystyczna– uracyl (U) na końcu 5’ i metylowany koniec 3’. Istnieją inne różnice:
- W przeciwieństwie do siRNA i mikroRNA nie wymagają one przetwarzania przez Dicer;
- Geny piRNA są aktywne tylko w komórkach rozrodczych (podczas embriogenezy) i otaczających je komórkach śródbłonka;
- Skład białek układu piRNA jest różny – są to endonukleazy klasy Piwi (Piwi i Aub) oraz osobna odmiana Argonaute – Ago3.
Przetwarzanie i aktywność piRNA są nadal słabo poznane, ale już wiadomo, że mechanizm działania jest zupełnie inny niż w przypadku innych krótkich RNA – dziś zaproponowano ping-pongowy model ich działania (ryc. 5 A, B).
Mechanizm ping-ponga biogenezy piRNA

Ryż. 5A: Cytoplazmatyczna część przetwarzania piRNA
W biogenezie i aktywności piRNA pośredniczy rodzina endonukleaz Piwi (Ago3, Aub, Piwi). Aktywność piRNA zapewniają zarówno jednoniciowe cząsteczki piRNA – sensowne, jak i antysensowne – z których każda wiąże się ze specyficzną endonukleazą Piwi. piRNA rozpoznaje komplementarny region mRNA transpozonu (niebieska nić) i wycina go. To nie tylko inaktywuje transpozon, ale także tworzy nowy piRNA (połączony z Ago3 poprzez metylację końca 3' przez metylazę Hen1). Ten piRNA z kolei rozpoznaje mRNA z transkryptami z klastra prekursorowego piRNA (nić czerwona) – w ten sposób cykl zostaje zamknięty i ponownie wytwarzany jest pożądany piRNA.

Ryż. 5B: piRNA w jądrze
Oprócz endonukleazy Aub, endonukleaza Piwi może również wiązać antysensowny piRNA. Po związaniu kompleks migruje do jądra, gdzie powoduje degradację komplementarnych transkryptów i przegrupowanie chromatyny, powodując supresję aktywności transpozonów.
Funkcje piRNA
Główną funkcją piRNA jest tłumienie aktywności MGE na poziomie transkrypcji i translacji. Uważa się, że piRNA są aktywne jedynie w okresie embriogenezy, kiedy nieprzewidywalne przetasowanie genomu jest szczególnie niebezpieczne i może doprowadzić do śmierci zarodka. To logiczne – kiedy układ odpornościowy jeszcze nie zaczął działać, komórki zarodka potrzebują prostej, ale skutecznej ochrony. Zarodek jest niezawodnie chroniony przed zewnętrznymi patogenami przez łożysko (lub skorupę jaja). Ale oprócz tego konieczna jest także obrona przed wirusami endogennymi (wewnętrznymi), przede wszystkim MGE.
Ta rola piRNA została potwierdzona doświadczeniem – „knockout”, czyli mutacje genów Ago3, Piwi czy Aub prowadzą do poważnych zaburzeń rozwojowych (i gwałtownego wzrostu liczby mutacji w genomie takiego organizmu), a także powodują niepłodność z powodu zakłócenia rozwoju komórek rozrodczych.
Dystrybucja i ewolucja piRNA
Pierwsze piRNA znaleziono już w ukwiałach i gąbkach. Rośliny najwyraźniej poszły inną drogą – nie znaleziono w nich białek Piwi, a rolę „kagańca” dla transpozonów pełni endonukleaza Ago4 i siRNA.
U zwierząt wyższych, w tym u ludzi, system piRNA jest bardzo dobrze rozwinięty, ale można go znaleźć tylko w komórkach embrionalnych i śródbłonku owodniowym. Czas pokaże, dlaczego dystrybucja piRNA w organizmie jest tak ograniczona. Można założyć, że jak każda potężna broń, piRNA przynoszą korzyść tylko w bardzo specyficznych warunkach (podczas rozwoju płodu), a w dorosłym organizmie ich działanie przyniesie więcej szkody niż pożytku. Mimo to liczba piRNA przekracza liczbę znanych białek o rząd wielkości, a niespecyficzne działanie piRNA w dojrzałych komórkach jest trudne do przewidzenia.
| siRNA | mikroRNA | piRNA | |
| Rozpościerający się | Rośliny, Drosophila, C. elegancja. Nie występuje u kręgowców | Eukarionty | Komórki embrionalne zwierząt (począwszy od koelenteratów). Nie u pierwotniaków i roślin |
| Długość | 21-22 nukleotydów | 19-25 nukleotydów | 24-30 nukleotydów |
| Struktura | Dwuniciowy, 19 komplementarnych nukleotydów i dwa niesparowane nukleotydy na końcu 3’ | Jednołańcuchowa złożona struktura | Jednołańcuchowa złożona struktura. U na końcu 5', 2'- O-metylowany koniec 3' |
| Przetwarzanie | Zależne od Dicera | Zależne od Dicera | Niezależny od Dicera |
| Endonukleazy | temu2 | temu1, temu2 | temu3, Piwi, Aub |
| Działalność | Degradacja komplementarnych mRNA, acetylacja genomowego DNA | Degradacja lub hamowanie translacji docelowego mRNA | Degradacja mRNA kodującego MGE, regulacja transkrypcji MGE |
| Rola biologiczna | Przeciwwirusowa obrona immunologiczna, tłumienie aktywności własnych genów | Regulacja aktywności genów | Tłumienie aktywności MGE podczas embriogenezy |
Wniosek
Podsumowując, chciałbym przedstawić tabelę ilustrującą ewolucję aparatu białkowego biorącego udział w interferencji RNA (ryc. 6). Można zauważyć, że pierwotniaki mają najbardziej rozwinięty system siRNA (rodziny białek Ago, Dicer), a w miarę jak organizmy stają się bardziej złożone, nacisk przesuwa się na układy bardziej wyspecjalizowane – liczbę izoform białek dla mikroRNA (Drosha, Pasha) i piRNA ( Piwi, Hen1) wzrasta. Jednocześnie zmniejsza się różnorodność enzymów pośredniczących w działaniu siRNA.

Ryż. 6. Różnorodność białek biorących udział w interferencji RNA
I
Liczby wskazują liczbę białek w każdej grupie. Elementy charakterystyczne dla siRNA i mikroRNA zaznaczono na niebiesko, a białka związane z piRNA na czerwono. Według .
Zjawisko interferencji RNA zaczęły wykorzystywać najprostsze organizmy. W oparciu o ten mechanizm natura stworzyła prototyp układu odpornościowego, a w miarę jak organizmy stają się coraz bardziej złożone, interferencja RNA staje się niezbędnym regulatorem aktywności genomu. Dwa różne mechanizmy plus trzy typy krótkich RNA (patrz tabela podsumowująca) – w rezultacie widzimy tysiące drobnych regulatorów różnych szlaków metabolicznych i genetycznych. Ten uderzający obraz ilustruje wszechstronność i ewolucyjną adaptację systemów biologii molekularnej. Krótkie RNA po raz kolejny udowadniają, że we wnętrzu komórki nie ma „małych rzeczy” – są jedynie małe cząsteczki, których pełne znaczenie dopiero zaczynamy rozumieć.
To prawda, że tak fantastyczna złożoność sugeruje raczej, że ewolucja jest „ślepa” i działa bez z góry zatwierdzonego „ plan główny» .
Literatura
- Gurdon J. B., Lane CD, Woodland HR, Marbaix G. (1971). Zastosowanie żabich jaj i oocytów do badania informacyjnego RNA i jego translacji w żywych komórkach. Natura 233, 177-182;
- Spirin AS (2001). Biosynteza białek, świat RNA i pochodzenie życia. Biuletyn Rosyjskiej Akademii Nauk 71, 320-328;
- Elementy: „Z sierści można teraz wyodrębnić kompletne genomy mitochondrialne wymarłych zwierząt”;
- Fire A., Xu S., Montgomery M.K., Kostas S.A., Driver S.E., Mello C.C. (1998). Silna i specyficzna interferencja genetyczna ze strony dwuniciowego RNA Caenorhabditis eleganckie. Natura 391, 806-311;
- Biomolekuła: „MikroRNA odkryte po raz pierwszy w organizmie jednokomórkowym”;
- Covey S., Al-Kaff N., Lángara A., Turner D. (1997). Rośliny zwalczają infekcję poprzez wyciszanie genów. Natura 385, 781-782;
- Biomolekuła: „Podwójne działanie molekularne: ludzkie geny działają na rzecz wirusa grypy”;
- Ren B. (2010). Transkrypcja: Wzmacniacze tworzą niekodujący RNA. Natura 465, 173–174;
- Taganov K.D., Boldin MP, Chang K.J., Baltimore D. (2006). Zależna od NF-κB indukcja mikroRNA miR-146, inhibitora ukierunkowanego na białka sygnalizacyjne wrodzonej odpowiedzi immunologicznej. Proc. Natl. Acad. Nauka. USA. 103, 12481-12486;
- O'Connell R. M., Rao D. S., Chaudhuri A. A., Boldin MP, Taganov K. D., Nicoll J., Paquette R. L., Baltimore D. (2008). Długotrwała ekspresja mikroRNA-155 w hematopoetycznych komórkach macierzystych powoduje zaburzenie mieloproliferacyjne. J. Exp. Med. 205, 585-594;
- Biomolekuła: „mikroRNA – im dalej w las, tym więcej drewna na opał”;
- Elementy: „Powikłania w organizmie starożytnych zwierząt wiązały się z pojawieniem się nowych cząsteczek regulatorowych”;
- Grimson A., Srivastava M., Fahey B., Woodcroft B.J., Chiang H.R., King N., Degnan B.M., Rokhsar D.S., Bartel D.P. (2008). Wczesne pochodzenie i ewolucja mikroRNA i RNA oddziałujących z Piwi u zwierząt. Natura 455, 1193–1197.
- Aravin A., Hannon G., Brennecke J. (2007). Ścieżka Piwi-piRNA zapewnia obronę adaptacyjną w wyścigu zbrojeń transpozonowych. Nauka 318, 761–764;
- Biocząsteczka: "
Artykuł na konkurs „bio/mol/text”: W ostatnich latach RNA – a zwłaszcza jego „nieklasyczne” odmiany – przyciąga uwagę biologów na całym świecie. Okazało się, że regulacja przez niekodujące RNA jest powszechna – od wirusów i bakterii po ludzi. Badanie różnorodności regulatorów małego bakteryjnego RNA wyraźnie wykazało ich ważną rolę zarówno w metabolizmie pośrednim, jak i odpowiedziach adaptacyjnych. W artykule opisano rodzaje małych RNA bakterii oraz mechanizmy regulacyjne realizowane za ich pomocą. Szczególny nacisk położony jest na rolę tych cząsteczek w życiu czynników bakteryjnych powodujących szczególnie niebezpieczne infekcje.
RNA: więcej niż tylko kopia DNA
Większość czytelników tej witryny zna podstawowe mechanizmy żywej komórki od czasów szkolnych. Na kursach biologii, od praw Mendla po najnowocześniejsze projekty sekwencjonowania genomu, czerwona nić przewija się przez ideę głównego programu genetycznego na rzecz rozwoju organizmu, znanego zawodowym biologom jako centralny dogmat biologii molekularnej. Stwierdza ona, że cząsteczka DNA pełni rolę nośnika i strażnika informacji genetycznej, która poprzez pośrednika – informacyjny RNA (mRNA) oraz przy udziale rybosomalnego (rRNA) i transferowego RNA (tRNA) – realizowana jest w postaci białek. Te ostatnie określają gatunek i indywidualny fenotyp.
Ten stan rzeczy i przypisanie RNA roli drugorzędnego uczestnika molekularnego funkcjonowania utrzymał się w środowisku naukowym aż do lat 80-tych ubiegłego wieku. Praca T. Cheka, który pokazał, że RNA może działać jak katalizator, zmusiła nas do bliższego przyjrzenia się RNA reakcje chemiczne. Wcześniej uważano, że przyspieszenie procesów chemicznych w komórce jest przywilejem enzymów, które mają wyłącznie charakter białkowy. Odkrycie aktywności katalitycznej w RNA miało daleko idące konsekwencje – podobnie jak wcześniejsze prace teoretyczne K. Woese’a i pozwoliło nakreślić możliwy obraz ewolucji prebiotycznej na naszej planecie. Faktem jest, że od chwili odkrycia funkcji DNA jako nośnika informacji genetycznej dylemat, co pojawiło się wcześniej w toku ewolucji – DNA czy białko niezbędne do reprodukcji DNA – wydawał się niemal równie filozoficzny (czyli bezsensowny) jako pytanie o prymat wyglądu kury czy jajka. Po odkryciu T. Cheka rozwiązanie nabrało bardzo realnego kształtu - odkryto cząsteczkę, która miała właściwości zarówno nośnika informacji, jak i biokatalizatora (aczkolwiek w swojej szczątkowej formie). Z biegiem czasu badania te rozrosły się do całego kierunku w biologii, badającego pochodzenie życia przez pryzmat tzw. „świata RNA”.
Stało się więc oczywiste świat starożytny RNA można powiązać z pochodzeniem i rozkwitem życia pierwotnego. Nie wynika jednak automatycznie z tego, że RNA we współczesnych organizmach nie jest archaizmem dostosowanym do potrzeb wewnątrzkomórkowych układów molekularnych, ale naprawdę ważnym uczestnikiem molekularnego zespołu komórki. Tylko rozwój metod molekularnych - w szczególności sekwencjonowania kwasy nukleinowe, - wykazało, że RNA jest naprawdę niezbędny w komórce, a nie tylko w postaci kanonicznej trójcy „mRNA, rRNA, tRNA”. Już pierwsze obszerne dane dotyczące sekwencjonowania DNA wskazywały na fakt, który początkowo wydawał się trudny do wyjaśnienia – większość z nich okazała się niekodujące- czyli nie niosący informacji o cząsteczkach białka lub „standardowym” RNA. Oczywiście można to częściowo przypisać „resztom genetycznym” – „wyłączonym” lub utraconym funkcjom fragmentów genomu. Jednak oszczędzanie takiej ilości „posagu” na systemy biologiczne, które starają się oszczędzać energię, wydaje się nielogiczne.
Rzeczywiście, bardziej szczegółowe i subtelne metody badawcze pozwoliły odkryć całą klasę RNA regulatorów ekspresji genów, częściowo wypełniających przestrzeń międzygenową. Jeszcze przed odczytaniem pełnych sekwencji genomów eukariotycznych glisty C. elegancja Wyizolowano mikroRNA – małe cząsteczki (około 20 nukleotydów), które mogą specyficznie wiązać się z regionami mRNA zgodnie z zasadą komplementarności. Łatwo się domyślić, że w takich przypadkach nie da się już odczytać informacji o zakodowanych białkach za pomocą mRNA: rybosom po prostu nie może „przebiec” przez takie miejsce, które nagle stało się dwuniciowe. Ten mechanizm tłumienia ekspresji genów, tzw Interferencja RNA, został już wystarczająco szczegółowo przeanalizowany na temat „biocząsteczki”. Do chwili obecnej odkryto tysiące cząsteczek mikroRNA i innych niekodujących RNA (piRNA, snoRNA, nanoRNA itp.). U eukariontów (w tym ludzi) znajdują się one w regionach międzygenowych. Ustalono ich ważną rolę w różnicowaniu komórek, karcynogenezie, odpowiedzi immunologicznej oraz innych procesach i patologiach.
Małe RNA są końmi trojańskimi dla białek bakteryjnych
Pomimo tego, że w bakteriach odkryto wiele niekodujących białek RNA wcześniej niż pierwszy podobnych regulatorów u eukariontów, ich rola w metabolizmie komórek bakteryjnych była od dawna zakryta dla społeczności naukowej. Jest to zrozumiałe – tradycyjnie komórkę bakteryjną uważano dla badacza za strukturę bardziej prymitywną i mniej tajemniczą, której złożoności nie da się porównać z nagromadzeniem struktur w komórce eukariotycznej. Ponadto w genomach bakteryjnych zawartość informacji niekodującej stanowi zaledwie kilka procent całkowitej długości DNA, a u niektórych prątków sięga maksymalnie 40%. Biorąc jednak pod uwagę, że mikroRNA występują nawet w wirusach, w bakteriach powinny one odgrywać ważną rolę regulacyjną, tym bardziej.
Okazało się, że prokarioty mają całkiem sporo małych regulatorów RNA. Tradycyjnie wszystkie można podzielić na dwie grupy:
- Cząsteczki RNA, które muszą wiązać się z białkami, aby spełniać swoją funkcję.
- RNA, które wiążą się komplementarnie z innymi RNA (zawierają większość znanych cząsteczek regulatorowych RNA).
Do pierwszej grupy zaliczają się małe RNA, dla których wiązanie z białkami jest możliwe, ale nie konieczne. Dobrze znanym przykładem jest RNaza P, która działa jak rybozym na „dojrzewającym” tRNA. Jeśli jednak RNaza P może funkcjonować bez składnika białkowego, to w przypadku innych małych RNA z tej grupy wiązanie z białkiem jest obowiązkowe (a one same są w rzeczywistości kofaktorami). Na przykład tmRNA aktywuje złożony kompleks białkowy, pełniąc rolę „klucza głównego” dla „zablokowanego” rybosomu – jeśli informacyjny RNA, z którego jest odczytywany, osiągnął swój koniec i nie natrafiono na kodon stop.
Znany jest również jeszcze bardziej intrygujący mechanizm bezpośredniego oddziaływania małych RNA z białkami. Białka wiążące się z „tradycyjnymi” kwasami nukleinowymi są szeroko rozpowszechnione w każdej komórce. Komórka prokariotyczna nie jest wyjątkiem. Na przykład jego białka histonopodobne pomagają prawidłowo upakować nić DNA, a specyficzne białka represorowe mają powinowactwo do regionu operatorowego genów bakteryjnych. Wykazano, że represory te mogą być hamowane przez małe RNA imitujące miejsca wiązania DNA „natywne” dla tych białek. Zatem na małym RNA CsrB (ryc. 1) znajduje się 18 miejsc „wabików”, które służą zapobieganiu dotarciu białka represora CsrA do jego prawdziwego celu – operonu glikogenu. Nawiasem mówiąc, wśród białek represorowych traconych pod wpływem tak małych RNA znajdują się regulatory globalnych szlaków metabolicznych, które umożliwiają wielokrotne wzmacnianie sygnału hamującego małego RNA. Na przykład to robi mały RNA 6S, „imitujący” współczynnik białkowy σ 70. Poprzez „oszustwo” konfiguracyjne, zajmując centra wiązania polimerazy RNA z czynnikiem sigma, zabrania ekspresji genów „uporządkowania”.
Rycina 1. Przewidywana bioinformatycznie struktura drugorzędowa małego RNA CsrB z Vibrio cholera M66-2. Małe RNA są cząsteczkami jednoniciowymi, ale podobnie jak inne RNA, składaniu się w stabilną strukturę przestrzenną towarzyszy tworzenie obszarów, w których cząsteczka hybrydyzuje ze sobą. Nazywa się liczne zagięcia konstrukcji w postaci otwartych pierścieni szpilki. W niektórych przypadkach kombinacja spinek do włosów pozwala RNA działać jak „gąbka”, niekowalencyjnie wiążąc określone białka. Częściej jednak cząsteczki tego typu zakłócają DNA lub RNA; w tym przypadku struktura przestrzenna małego RNA zostaje zakłócona i powstają nowe miejsca hybrydyzacji z cząsteczką docelową. Mapa cieplna odzwierciedla prawdopodobieństwo, że odpowiednia para nukleotydów zostanie faktycznie połączona wewnątrzcząsteczkowym wiązaniem wodorowym; dla sekcji niesparowanych - prawdopodobieństwo utworzenia wiązań wodorowych z dowolnymi sekcjami wewnątrz cząsteczki. Obraz uzyskano za pomocą programu RNAfold.
Małe RNA bakterii zakłócają... i to bardzo skutecznie!
Mechanizm działania regulatorów drugiej grupy jest na ogół podobny do regulatorowego RNA u eukariontów – jest to ta sama interferencja RNA poprzez hybrydyzację z mRNA, tyle że same łańcuchy małych RNA są często dłuższe – nawet do kilkuset nukleotydy ( cm. Ryż. 1). W rezultacie, ze względu na mały RNA, rybosomy nie mogą odczytać informacji z mRNA. Choć często, jak się wydaje, do tego nie dochodzi: powstałe kompleksy „mały RNA – mRNA” stają się celem RNaz (takich jak RNaza P).
Zwartość i gęstość upakowania genomu prokariotycznego daje się odczuć: jeśli u eukariontów większość regulatorowych RNA jest zapisana w oddzielnych (najczęściej niekodujących białka) loci, wówczas wiele małych RNA bakterii można zakodować w tym samym regionie DNA, co tłumiony gen, ale na przeciwległych łańcuchach! Te RNA nazywane są kodowany w formacie cis(antysensowny) i małe RNA leżące w pewnej odległości od tłumionej części DNA - transkodowane. Najwyraźniej ułożenie cis-RNA można uznać za triumf ergonomii: można je odczytać z przeciwnej nici DNA w momencie jej rozwinięcia jednocześnie z docelowym transkryptem, co pozwala na precyzyjną kontrolę ilości syntetyzowanego białka.
Małe RNA w trans ewoluują niezależnie od docelowego mRNA, a sekwencja regulatora zmienia się silniej w wyniku mutacji. Być może ta sytuacja jest korzystna tylko dla komórki bakteryjnej, ponieważ mały RNA nabiera aktywności wobec wcześniej nietypowych celów, co zmniejsza czas i koszty energii potrzebne na tworzenie innych regulatorów. Z drugiej strony presja selekcyjna zapobiega nadmiernej mutacji trans małego RNA, ponieważ utraci on aktywność. Jednakże, aby hybrydyzować z informacyjnym RNA, większość trans-małych RNA wymaga elementu pomocniczego, białka Hfq. W przeciwnym razie niepełna komplementarność małego RNA może powodować problemy z wiązaniem z celem.
Najwyraźniej potencjalny mechanizm regulacyjny oparty na zasadzie „jeden mały RNA – wiele celów” pomaga zintegrować sieci metaboliczne bakterii, co jest niezwykle niezbędne w warunkach krótkiego życia pojedynczej komórki. Można kontynuować spekulacje na ten temat i założyć, że za pomocą transkodowanych małych RNA „instrukcje” ekspresyjne są wysyłane z funkcjonalnie powiązanych, ale fizycznie odległych loci. Logicznie wyjaśniono potrzebę tego rodzaju genetycznego „apelu”. duża liczba małe RNA występujące w bakteriach chorobotwórczych. Na przykład kilkaset małych RNA znaleziono u rekordzisty tego wskaźnika - Vibrio cholerae ( Vibrio cholera). To mikroorganizm, który może przetrwać w otaczającym środowisku wodnym (zarówno świeżym, jak i słonym), a także na wodnych skorupiakach, rybach i jelitach człowieka - nie da się obejść bez kompleksowej adaptacji za pomocą cząsteczek regulatorowych!
CRISPR chroni zdrowie bakterii
Małe RNA wykorzystano także do rozwiązania innego palącego problemu bakterii. Nawet najbardziej złośliwe patogenne ziarniaki i prątki mogą być bezsilne w obliczu zagrożenia, jakie stwarzają specjalne wirusy - bakteriofagi, zdolne do błyskawicznego niszczenia populacji bakterii. Organizmy wielokomórkowe posiadają wyspecjalizowany system ochrony przed wirusami - odporny za pomocą komórek i wydzielanych przez nie substancji chroniących organizm nieproszeni goście(w tym o charakterze wirusowym). Komórka bakteryjna jest samotnikiem, ale nie jest tak bezbronna, jak mogłoby się wydawać na pierwszy rzut oka. Loci pełnią rolę strażników receptur na utrzymanie przeciwwirusowej odporności bakterii CRISPR- skupione, regularnie przerywane krótkie powtórzenia palindromiczne ( skupione, regularnie przeplatane krótkimi powtórzeniami palindromowymi) (ryc. 2; ). W genomach prokariotycznych każda kaseta CRISPR jest reprezentowana przez sekwencję liderową o długości kilkuset nukleotydów, po której następuje seria 2–24 (czasami do 400) powtórzeń oddzielonych regionami rozdzielającymi o podobnej długości, ale unikalnej sekwencji nukleotydów. Długość każdego odstępnika i powtórzenia nie przekracza stu par zasad.

Rycina 2. Locus CRISPR i przetwarzanie odpowiadającego mu małego RNA w funkcjonalny transkrypt. W genomie CRISPR- kasetę reprezentują przeplatające się ze sobą przekładki (na rysunku są oznaczone jako Sp), częściowo homologiczny do regionów DNA faga i powtórzeń ( Przez) o długości 24–48 pz, co świadczy o symetrii diadycznej. W przeciwieństwie do powtórzeń, odstępniki w tym samym locus mają tę samą długość (u różnych bakterii może to być 20–70 nukleotydów), ale różnią się sekwencją nukleotydów. Sekcje „powtórzenia odstępu” mogą być dość długie i składać się z kilkuset jednostek. Cała struktura jest otoczona z jednej strony sekwencją liderów ( płyta długogrająca, kilkaset par zasad). Geny Cas znajdują się w pobliżu ( C RISPR-Jak powiązane), zorganizowane w operę. Odczytane z nich białka pełnią szereg funkcji pomocniczych, zapewniając obróbkę odczytanego transkryptu CRISPR-locus, jego udana hybrydyzacja z docelowym DNA faga, wstawienie nowych elementów do locus, itp. CrRNA powstały w wyniku wieloetapowej obróbki hybrydyzuje z odcinkiem DNA (dolna część rysunku) wstrzykniętym przez faga do bakterii. To wycisza maszynę transkrypcyjną wirusa i zatrzymuje jego reprodukcję w komórce prokariotycznej.
Szczegółowy mechanizm powstawania wszystkiego CRISPR-locus pozostaje do zbadania. Ale dzisiaj zaproponowano schematyczny diagram wyglądu przekładek, najważniejszych struktur w jego składzie. Okazuje się, że „łowców bakterii” bije ich własna broń – kwasy nukleinowe, a raczej „trofeum” informacji genetycznej otrzymanej przez bakterie od fagów w poprzednich bitwach! Faktem jest, że nie wszystkie fagi, które dostaną się do komórki bakteryjnej, okazują się śmiertelne. DNA takich fagów (prawdopodobnie sklasyfikowanych jako umiarkowane) jest cięty przez specjalne białka Cas (ich geny flankują CRISPR) na małe fragmenty. Niektóre z tych fragmentów zostaną osadzone w CRISPR- loci genomu „gospodarza”. A kiedy DNA faga ponownie dostanie się do komórki bakteryjnej, napotyka mały RNA CRISPR-locus, w tym momencie wyrażany i przetwarzany przez białka Cas. Następnie następuje inaktywacja informacji genetycznej wirusa, zgodnie z mechanizmem interferencji RNA opisanym już powyżej.
Z hipotezy tworzenia przekładek nie jest jasne, dlaczego potrzebne są między nimi powtórzenia, w obrębie jednego miejsca o nieco różnej długości, ale prawie identycznej sekwencji? Otwiera się tutaj szeroko otwarta przestrzeń dla fantazji. Być może bez powtórzeń problematyczne byłoby podzielenie danych genetycznych na fragmenty semantyczne, przypominające sektory na dysku twardym komputera, a następnie dostęp maszyny transkrypcyjnej do ściśle określonych obszarów CRISPR-locus stałoby się trudne? A może powtórzenia upraszczają procesy rekombinacji po wstawieniu nowych elementów DNA faga? A może są to „znaki interpunkcyjne” niezbędne do przetwarzania CRISPR? Tak czy inaczej, w odpowiednim czasie zostanie odnaleziona biologiczna przyczyna wyjaśniająca zachowanie komórki bakteryjnej na wzór Pliuszkina Gogola.
CRISPR, będący „kroniką” związku bakterii z fagiem, może znaleźć zastosowanie w badaniach filogenetycznych. Zatem ostatnio przeprowadzono typowanie wg CRISPR pozwoliło nam przyjrzeć się ewolucji poszczególnych szczepów drobnoustroju dżumy ( Yersinia pestis). Zbadaj je CRISPR- „rodowody” rzuciły światło na wydarzenia sprzed pół tysiąca lat, kiedy szczepy przybyły do Mongolii z terenów dzisiejszych Chin. Ale nie dla wszystkich bakterii, a w szczególności patogenów, Ta metoda odpowiedni. Pomimo niedawnych dowodów wskazujących na przewidywaną obróbkę białek CRISPR u patogenów tularemii ( Francisella tularensis) i cholerę, same CRISPR, jeśli są obecne w ich genomie, są nieliczne. Być może fagi, biorąc pod uwagę ich pozytywny wkład w nabywanie zjadliwości przez patogennych przedstawicieli królestwa bakterii, nie są na tyle szkodliwe i niebezpieczne, aby się przed nimi bronić za pomocą CRISPR? A może wirusy atakujące te bakterie są zbyt różnorodne i strategia „zakłócania” odporności na RNA przeciwko nim jest daremna?

Rysunek 3. Niektóre mechanizmy działania ryboprzełącznika. Ryboprzełączniki (ryboprzełączniki) są wbudowane w informacyjny RNA, ale wyróżniają się dużą swobodą zachowań konformacyjnych w zależności od konkretnych ligandów, co daje podstawy do uznania ryboprzełączników za niezależne jednostki małych RNA. Zmiana konformacji platformy ekspresyjnej wpływa na miejsce lądowania rybosomu w mRNA ( RBS), a w konsekwencji określa dostępność całego mRNA do odczytu. Ryboswitchy przypominają w pewnym stopniu domenę operatorską w modelu klasycznym gumilaka-operon - ale tylko regiony aptamerów są zwykle regulowane przez substancje niskocząsteczkowe i przełączają działanie genów na poziomie mRNA, a nie DNA. A - W przypadku braku ligandów, ryboprzełączniki btuB (transporter kobalaminy) I tiM (zależny od pirofosforanu tiaminy), które przeprowadzają nienukleolityczną represję mRNA, są „włączone” ( NA) i pozwalają rybosomowi zająć się swoimi sprawami. Wiązanie ligandu z ryboprzełącznikiem ( WYŁĄCZONY-pozycja) prowadzi do powstania spinki do włosów, czyniąc ten obszar niedostępnym dla rybosomu. B - Ryboprzełącznik lizynowy lysC w przypadku braku ligandu jest również uwzględniony ( NA). Wyłączenie przełącznika rybosomowego blokuje dostęp rybosomu do mRNA. Ale w przeciwieństwie do opisanych powyżej ryboprzełączników, w przełączniku lizynowym, po wyłączeniu, sekcja zostaje „odsłonięta”, przecięta specjalnym kompleksem RNazy ( degradosom) i cały mRNA jest wykorzystywany, rozkładając się na małe fragmenty. Represja przez ryboswitch w tym przypadku nazywana jest nukleolityczną ( nukleolityczny) i jest nieodwracalny, ponieważ w odróżnieniu od przykładu ( A ), przełączanie wsteczne (powrót do NA) nie jest już możliwe. Należy zauważyć, że w ten sposób można osiągnąć wykorzystanie grupy „niepotrzebnych” mRNA: ryboswitch przypomina część zestawu konstrukcyjnego dla dzieci, a cała grupa funkcjonalnie powiązanych cząsteczek matrix może mieć przełączniki o podobnej budowie Struktura.
Riboswitch - czujnik bakterii
Istnieją zatem małe RNA powiązane z białkami, małe RNA zakłócające własne mRNA bakterii, a także RNA wychwytywane przez bakterie z wirusów i tłumiące DNA faga. Czy można sobie wyobrazić inny mechanizm regulacji przy użyciu małych RNA? Okazuje się, że tak. Jeśli przeanalizujemy to, co opisano powyżej, odkryjemy, że we wszystkich przypadkach regulacji antysensownej, interferencję małego RNA z obiektem docelowym obserwuje się w wyniku hybrydyzacji dwóch indywidualny Cząsteczki. Dlaczego nie uporządkować małego RNA jako część samego transkrypcji? Wówczas możliwa jest zmiana konformacji takiego „zagubionego kozaka” wewnątrz mRNA, zmiana dostępności całego szablonu do odczytu podczas translacji lub, co jest jeszcze bardziej celowe energetycznie, regulacja biosyntezy mRNA, tj. transkrypcja!
Struktury takie są szeroko obecne w komórkach bakteryjnych i są znane jako ryboprzełączniki ( ryboprzełącznik). Znajdują się one przed początkiem części kodującej genu, na końcu 5' mRNA. Konwencjonalnie w składzie ryboprzełączników można wyróżnić dwa motywy strukturalne: region aptameru, odpowiedzialny za wiązanie z ligandem (efektorem) i platforma ekspresyjna, zapewniając regulację ekspresji genów poprzez przejście mRNA do alternatywnych struktur przestrzennych. Do działania służy np. taki wyłącznik (typu „wyłączony”) operon lizynowy: gdy jest nadmiar lizyny, występuje ona w postaci „splątanej” strukturę przestrzenną, co blokuje odczyt z operonu, a w przypadku jego braku następuje „rozwinięcie” ryboswitcha i synteza białek niezbędnych do biosyntezy lizyny (ryc. 3).
Opisany schemat urządzenia ryboswitch nie jest kanoniczny, istnieją odmiany. U Vibrio cholerae odkryto ciekawy „włączający” tandemowy przełącznik rybosowy: platformę ekspresyjną poprzedza dwa na raz region aptameru. Zapewnia to oczywiście większą czułość i płynniejszą reakcję na pojawienie się w komórce innego aminokwasu – glicyny. Być może „podwójny” przełącznik ryboprzełącznikowy w genomie patogenu, podobny w zasadzie działania, pośrednio wpływa na wysoką przeżywalność bakterii wąglik (Bacillus anthracis). Reaguje ze związkiem wchodzącym w skład pożywki minimalnej i niezbędnym dla tego drobnoustroju – pirofosforanem tiaminy.
Oprócz przełączania szlaków metabolicznych w zależności od „menu” dostępnego komórce bakteryjnej, ryboswitchy mogą być czujnikami homeostazy bakteryjnej. Tym samym dostrzeżono je w regulacji dostępności genu do odczytu, gdy funkcjonowanie układu translacyjnego wewnątrz komórki zostaje zakłócone (np. sygnały takie jak pojawienie się „nienaładowanych” tRNA i „wadliwych” (zatrzymanych) rybosomów ) lub gdy zmieniają się czynniki środowiskowe (na przykład wzrost temperatury).
Nie potrzeba białek, daj nam RNA!
Co zatem oznacza obecność tak dużej różnorodności regulatorów małego RNA wewnątrz bakterii? Czy oznacza to odrzucenie koncepcji, w której głównymi „menedżerami” są białka, czy też mamy do czynienia z kolejnym trendem w modzie? Widocznie ani jedno, ani drugie. Oczywiście niektóre małe RNA są globalnymi regulatorami szlaków metabolicznych, jak np. wspomniany CsrB, który wraz z CsrC bierze udział w regulacji magazynowania węgla organicznego. Biorąc jednak pod uwagę zasadę duplikacji funkcji w układach biologicznych, małe RNA bakterii można porównać raczej do „menedżera kryzysowego” niż do dyrektora generalnego. Tym samym w warunkach gdzie dla przetrwania mikroorganizmu jest to konieczne szybko rekonfigurują metabolizm wewnątrzkomórkowy, ich rola regulacyjna może być decydująca i skuteczniejsza niż białka o podobnych funkcjach. Zatem regulatory RNA odpowiadają raczej za szybką reakcję, mniej stabilną i niezawodną niż w przypadku białek: nie powinniśmy zapominać, że mały RNA zachowuje swoją strukturę 3D i jest utrzymywany na hamowanej matrycy przez słabe wiązania wodorowe.
Pośrednim potwierdzeniem tych tez mogą być wspomniane już małe RNA Vibrio cholerae. Dla tej bakterii wejście do organizmu ludzkiego nie jest pożądanym celem, ale najwyraźniej sytuacją awaryjną. Produkcja toksyn i aktywacja innych szlaków związanych z zjadliwością jest w tym przypadku jedynie reakcją obronną na agresywną opozycję środowiska i komórek organizmu wobec „obcych”. „Zbawicielami” są tutaj małe RNA, na przykład Qrr, które pomagają vibrio w stresujących warunkach modyfikować jego strategię przetrwania, zmieniając zbiorowe zachowanie. Hipotezę tę można również pośrednio potwierdzić odkryciem małego RNA VrrA, które jest aktywnie syntetyzowane, gdy w organizmie znajdują się wibratory i hamuje wytwarzanie białek błonowych Omp. „Ukryte” białka błonowe w początkowej fazie infekcji mogą pomóc uniknąć silnej odpowiedzi immunologicznej organizmu człowieka (ryc. 4).

Rycina 4. Małe RNA w realizacji właściwości patogennych Vibrio cholerae. A - Vibrio cholerae czuje się dobrze i dobrze rozmnaża się w środowisku wodnym. Organizm ludzki prawdopodobnie nie jest główną niszą ekologiczną dla tego drobnoustroju. B - Po przedostaniu się drogą wodną lub pokarmową zakażenia do agresywnego środowiska - jelita cienkiego człowieka - wibracja pod względem zorganizowanego zachowania zaczyna przypominać pseudoorganizm, którego głównym zadaniem jest powstrzymywanie odpowiedzi immunologicznej i stworzyć sprzyjające środowisko do kolonizacji. Bardzo ważne Pęcherzyki błonowe odpowiadają za koordynację działań w populacji bakterii i ich interakcję z organizmem. Nie do końca poznane czynniki środowiskowe w jelicie działają jako sygnały ekspresji małych RNA (na przykład VrrA) w wibriosach. W efekcie zostaje uruchomiony mechanizm powstawania pęcherzyków, które przy małej liczbie komórek Vibrio w jelicie są nieimmunogenne. Oprócz opisanego efektu, małe RNA pomagają „ukryć” białka błonowe Omp, które są potencjalnie prowokacyjne dla układu odpornościowego człowieka. Przy pośrednim udziale małych RNA Qrr1-4 uruchamiana jest intensywna produkcja toksyny cholery (niepokazanej na rysunku), co uzupełnia zakres reakcji adaptacyjnych Vibrio cholerae. V - W ciągu kilku godzin liczba komórek bakteryjnych wzrasta, a pula małych RNA VrrA maleje, co prawdopodobnie prowadzi do odsłonięcia białek błonowych. Stopniowo zmniejsza się także liczba „pustych” pęcherzyków, które na tym etapie są zastępowane immunogennymi, dostarczanymi do enterocytów. Najwyraźniej jest to część „planu” wdrożenia złożonego sygnału, którego znaczeniem jest sprowokowanie ewakuacji wibracji z ludzkiego ciała. Uwaga: nie obserwuje się stosunku wielkości komórek bakteryjnych do enterocytów.
Ciekawie będzie zobaczyć, jak zmieni się nasze rozumienie regulatorów małego RNA, gdy zostaną uzyskane nowe dane na platformach RNAseq, w tym na temat form wolno żyjących i niehodowanych. Niedawne prace z wykorzystaniem „głębokiego sekwencjonowania” przyniosły już nieoczekiwane wyniki, wskazując na obecność cząsteczek podobnych do mikroRNA u zmutowanych paciorkowców. Oczywiście takie dane wymagają dokładnego sprawdzenia, ale tak czy inaczej, możemy śmiało powiedzieć, że badanie małych RNA w bakteriach przyniesie wiele niespodzianek.
Podziękowanie
Oryginalne pomysły i projekt kompozycyjny podczas tworzenia obrazu tytułowego, a także zdjęcia 4, należą do absolwenta Instytutu Archiologii Południowego Uniwersytetu Federalnego Kopaeva E.A. Obecność ryciny 2 w artykule jest zasługą profesora nadzwyczajnego katedry. Zoologia SFU G.B. Bachtadze. Dokonał także korekty naukowej i rewizji ryciny tytułowej oraz ryciny 4. Autor wyraża im głęboką wdzięczność za cierpliwość i twórcze podejście do sprawy. Specjalne podziękowania kieruję do mojego kolegi, starszego badacza. laboratorium. biochemia drobnoustrojów Rostowskiego Instytutu Przeciw zarazie Sorokin V.M. za dyskusję nad tekstem artykułu i podzielenie się cennymi uwagami.
Literatura
- Carl Woese (1928–2012) ;;. 80 , 1148-1154;
- R. R. Breaker. (2012). Ryboswitchy i świat RNA. Perspektywy Cold Spring Harbor w biologii. 4 , a003566-a003566;
- J. Patrick Bardill, Brian K. Hammer. (2012). Niekodujące sRNA regulują zjadliwość patogenu bakteryjnego Vibrio cholerae. Biologia RNA. 9 , 392-401;
- Heon-Jin Lee, Su-Hyung Hong. (2012). Analiza małych RNA o wielkości mikroRNA u Streptococcus mutans metodą głębokiego sekwencjonowania. FEMS Microbiol Lett. 326 , 131-136;
- POSEŁ. Caron, L. Bastet, A. Lussier, M. Simoneau-Roy, E. Masse, DA Lafontaine. (2012). Przełącznik ryboswitchowy o podwójnym działaniu kontroluje inicjację translacji i rozpad mRNA. Postępowanie Narodowej Akademii Nauk. 109 , E3444-E3453.
JESTEM. Deichman, S.V. Zinowjew, A.Yu.Barysznikow
EKSPRESJA GENÓW I MAŁY RNA W ONKOLOGII
GU RONC im. N.N.Blokhin RAMS, Moskwa
STRESZCZENIE
W artykule przedstawiono rolę małych RNA kontrolujących większość funkcji życiowych komórki i organizmu oraz ich możliwy związek w szczególności z onkogenezą i innymi (w tym hipotetycznymi) wewnątrzkomórkowymi mechanizmami ekspresji genomu.
Słowa kluczowe: małe RNA, interferencja RNA (RNAi), dwuniciowy RNA (dsRNA), edycja RNA, onkogeneza.
JESTEM. Deichmana, S.V.Zinowiew, A.Yu.Baryshnikov.
EKSPRESJA GENÓW I MAŁYCH RNA W ONKOLOGII
N.N. Błochin Rosyjskie Centrum Badań nad Rakiem RAMS w Moskwieoch
ABSTRAKCYJNY
W artykule przedstawiono rolę małych RNA nadzorujących większość funkcji życiowych komórki i organizmu oraz możliwe ich powiązanie w szczególności z onkogenezą i innymi (w tym hipotetycznymi) wewnątrzkomórkowymi mechanizmami ekspresji genomu.
Słowa kluczowe: Małe RNA, interferencyjne RNA (RNAi), dwuniciowe RNA (dsRNA), edycja RNA, nowotworzenie.
Wstęp
Ekspresja poszczególnych genów i całych genomów eukariotycznych, obejmująca przetwarzanie, różne rodzaje transkrypcji, splicingu, rearanżacji, edycji RNA, rekombinacji, translacji, interferencji RNA, jest regulowana przez określone białka (produkty genów regulatorowych, strukturalnych, homeotycznych, czynniki transkrypcyjne). , elementy ruchome, RNA i efektory o niskiej masie cząsteczkowej. Do przetwarzających RNA zalicza się rRNA, tRNA, mRNA, niektóre typy regulatorowego RNA i małe RNA.
Obecnie wiadomo, że małe RNA nie kodują białek, często liczonych w setkach w genomie, i biorą udział w regulacji ekspresji różnych genów eukariotycznych (komórek somatycznych, immunologicznych, rozrodczych, macierzystych). Procesy różnicowania (hematopoeza, angiogeneza, adipogeneza, miogeneza, neurogeneza), morfogeneza (m.in. stadia embrionalne, rozwój/wzrost, regulacja fizjologiczna), proliferacja, apoptoza, karcynogeneza, mutageneza, immunogeneza, starzenie się (przedłużanie życia), wyciszanie epigenetyczne; odnotowano przypadki regulacji metabolicznej (na przykład glikosfingolipidów). Szerszą klasę niekodujących RNA o długości 20-300/500 nukleotydów i ich RNP można znaleźć nie tylko w jądrze/jąderku/cytoplazmie, ale także w organellach komórkowych zawierających DNA (mitochondria zwierzęce; mikroRNA i małe sekwencje konsensusowe dla transkryptów chloroplastów znaleziono w roślinnym RNA).
Do kontroli i regulacji v.n. W procesach ważne jest: 1. aby małe naturalne/sztuczne RNA (małe RNA, tRNA itp.) i ich kompleksy z białkami (RNP) były zdolne do przezbłonowego transportu komórkowego i mitochondrialnego; 2. że po rozpadzie mitochondriów część ich zawartości, RNA i RNP, może trafić do cytoplazmy i jądra. Wymienione właściwości małych RNA (SRNA), których funkcjonalnie istotna rola dopiero wzrasta w procesie badań, mają oczywiście związek z czynnikiem czujności w przypadku nowotworów i innych chorób genetycznych. Jednocześnie stało się jasne, jak duże znaczenie mają epigenomiczne modyfikacje chromatyny w występowaniu nowotworów. Rozważymy tylko bardzo ograniczoną liczbę przypadków spośród wielu podobnych.
Małe RNA
Mechanizm działania małych RNA polega na ich zdolności do niemal komplementarnego wiązania się z nieulegającymi translacji regionami 3” (3”-UTR) docelowych mRNA (które czasami zawierają elementy DNA/RNA transponujące MIR/LINE-2, a także konserwatywne Alu powtórzenia) i powodują interferencję RNA (RNAi=RNAi; w szczególności podczas odpowiedzi przeciwwirusowej). Komplikacją jest jednak to, że oprócz komórkowych istnieją również kodowane przez wirusy małe RNA (herpes, SV40 itp.; na przykład EBV zawiera 23, a KSHV - 12 miRNA), które oddziałują z transkryptami zarówno wirus i gospodarz. Znanych jest ponad 5 tysięcy samych miRNA komórkowych/wirusowych u 58 gatunków. RNAi inicjuje albo degradację (przy udziale kompleksu RISC, RNA-Induced Silencing Complex) wzdłuż wrażliwych na nukleazy fragmentów ciągłych helis lncRNA (dwuniciowy mRNA RNA itp.), albo częściowo odwracalne hamowanie nieciągłych helikalnych lncRNA podczas translacji docelowe mRNA. Dojrzałe małe RNA (~15-28 nukleotydów) powstają w cytoplazmie z ich prekursorów poddanych obróbce jądrowej o różnej długości (dziesiątki i setki nukleotydów). Ponadto małe RNA biorą udział w tworzeniu cichej struktury chromatyny, regulacji transkrypcji poszczególnych genów, tłumieniu ekspresji transpozonów i utrzymaniu struktury funkcjonalnej rozszerzonych regionów heterochromatyny.
Istnieje kilka głównych typów małych RNA. Najlepiej zbadane są mikroRNA (miRNA) i małe interferujące RNA (siRNA). Ponadto wśród małych RNA badane są: piRNA aktywne w komórkach rozrodczych; małe interferujące RNA związane z endogennymi retrotranspozonami i elementami powtarzalnymi (z lokalną/globalną heterochromatyzacją – począwszy od wczesnych etapów embriogenezy; utrzymują poziom telomerów), rasiRNA Drosophila; często kodowane przez introny genów białkowych i funkcjonalnie ważne w translacji, transkrypcji, splicingu (de-/metylacja, pseudourydylacja kwasów nukleinowych) małych jądrowych (snRNA) i jąderkowych (snoRNA) RNA; małe modulatory RNA, smRNA, o mało znanych funkcjach, komplementarne do wiążących DNA motywów NRSE (Neuron Restrictive Silencer Element); roślinne transaktywujące małe interferujące RNA, tasiRNA; krótkie RNA o strukturze spinki do włosów, shRNA, zapewniające długotrwałe RNAi (stabilne wyciszanie genów) długich struktur lncRNA podczas odpowiedzi przeciwwirusowej u zwierząt.
Małe RNA (miRNA, siRNA itp.) oddziałują z nowo zsyntetyzowanymi transkryptami jądra/cytoplazmy (regulując splicing, translację mRNA; metylacja/pseudourydylacja rRNA itp.) i chromatyną (podczas tymczasowej lokalnej i dziedziczonej epigenetycznie heterochromatynizacji dzielących się somatycznych komórki rozrodcze). W szczególności heterochromatynizacji towarzyszy de-/metylacja DNA, a także metylacja, acetylacja, fosforylacja i ubikwitynacja histonów (modyfikacja „kodu histonowego”).
Pierwszymi spośród małych RNA były miRNA nicienia Caenorhabditis elegans (lin-4), ich właściwości i geny, a nieco później miRNA rośliny Arabidopsis thaliana. Obecnie są z nimi związani Organizmy wielokomórkowe, chociaż wykazano je u jednokomórkowych alg Chlamydomonas reinhardtii oraz szlaki wyciszające podobne do RNAi, w powiązaniu z ochroną przeciwwirusową/podobną z udziałem tzw. psiRNA, omówione dla prokariotów. Genomy wielu eukariontów (w tym Drosophila i ludzi) zawierają kilkaset genów miRNA. Te geny specyficzne dla etapu/tkanki (jak również odpowiadające im docelowe regiony mRNA) są często wysoce homologiczne u gatunków odległych filogenetycznie, ale niektóre z nich są specyficzne dla linii. miRNA zawarte są w eksonach (kodujących białka, genach RNA), intronach (najczęściej pre-mRNA), odstępnikach międzygenowych (w tym powtórzeniach), mają długość do 70-120 nukleotydów (lub więcej) i tworzą pętlę/rdzeń spinki do włosów Struktury. Aby określić ich geny, stosuje się nie tylko podejście biochemiczne i genetyczne, ale także podejście komputerowe.
Najbardziej typowa długość „obszaru roboczego” dojrzałych miRNA wynosi 21–22 nukleotydów. Są to prawdopodobnie najliczniejsze geny niekodujące białek. Mogą one występować w postaci pojedynczych kopii (częściej) lub klastrów zawierających wiele podobnych lub różnych genów miRNA, transkrybowanych (często z promotorów autonomicznych) jako dłuższy prekursor, przetwarzanych w kilku etapach na pojedyncze miRNA. Uważa się, że istnieje sieć regulacyjna miRNA, która kontroluje wiele podstawowych procesów biologicznych (w tym powstawanie nowotworów/przerzuty); prawdopodobnie co najmniej 30% genów ulegających ekspresji u ludzi jest regulowanych przez miRNA.
W procesie tym biorą udział specyficzne dla lncRNA enzymy podobne do RNazy III Drosha (rybonukleaza jądrowa; inicjuje przetwarzanie intronowych pre-miRNA po splicowaniu głównego transkryptu) i Dicer, który działa w cytoplazmie i odpowiednio rozszczepia/rozszczepia pre-miRNA do włosów miRNA (do dojrzałych miRNA) i struktury hybrydowe miRNA/mRNA utworzone później. Małe RNA wraz z kilkoma białkami (w tym vn RNazami, białkami z rodziny AGO, transmetylazami/acetylazami itp.) oraz z udziałem tzw. Kompleksy typu RISC i RITS (drugi indukuje wyciszanie transkrypcji) są zdolne odpowiednio do powodowania/degradacji RNAi i późniejszego wyciszania genów na poziomach RNA (przed/w trakcie translacji) i DNA (podczas transkrypcji heterochromatyny).
Każdy miRNA potencjalnie łączy się w parę z wieloma celami, a każdy cel jest kontrolowany przez pewną liczbę miRNA (co przypomina edycję pre-mRNA za pośrednictwem gRNA w kinetoplastach trypanosomu). Analiza in vitro wykazała, że regulacja miRNA (jak również edycja RNA) jest kluczowym posttranskrypcyjnym modulatorem ekspresji genów. Podobne miRNA konkurujące o ten sam cel są potencjalnymi transregulatorami interakcji RNA-RNA i RNA-białko.
U zwierząt miRNA najlepiej badać u nicienia Caenorhabditis Elegans; opisano ponad 112 genów. Znaleziono tu także tysiące endogennych siRNA (brak genów; związanych w szczególności z transkryptami i transpozonami, w których pośredniczy spermatogeneza). Obydwa małe RNA metazoanów mogą być generowane przez polimerazy RNA, które wykazują aktywność (nie homologię) typów RdRP-II (jak większość innych RNA) i RdRP-III. Dojrzałe małe RNA mają podobny skład (w tym końcowe 5"-fosforany i 3"-OH), długość (zwykle 21-22 nukleotydów) i funkcję i mogą konkurować o ten sam cel. Jednakże degradacja RNA, nawet przy całkowitej komplementarności celu, jest częściej powiązana z siRNA; represja translacyjna, z częściową, zwykle 5-6 nukleotydami, komplementarność - z miRNA; a prekursory, odpowiednio, są egzo-/endogenne (setki/tysiące nukleotydów) dla siRNA i zwykle endogenne (dziesiątki/setki nukleotydów) dla miRNA, a ich biogeneza jest inna; jednakże w niektórych systemach różnice te są odwracalne.
RNAi, w którym pośredniczą siRNA i miRNA, pełni różnorodne naturalne role: od regulacji ekspresji genów i heterochromatyny po ochronę genomu przed transpozonami i wirusami; ale siRNA i niektóre miRNA nie są konserwatywne między gatunkami. W roślinach (Arabidopsis thaliana) stwierdzono: siRNA odpowiadające zarówno genom, jak i regionom międzygenowym (w tym przerywnikom, powtórzeniom); ogromną liczbę potencjalnych miejsc w genomie dla różnych typów małych RNA. Nicienie posiadają również tzw zmienne, wyrażane autonomicznie 21U-RNA (dasRNA); mają 5"-Y-monofosforan, zawierają 21 nukleotydów (20 z nich jest zmiennych) i są zlokalizowane pomiędzy lub wewnątrz intronów genów kodujących białka w ponad 5700 miejscach w dwóch regionach chromosomu IV.
MiRNA odgrywają ważną rolę w ekspresji genów w zdrowiu i chorobie; u człowieka występuje co najmniej 450-500 takich genów. Zwykle wiążąc się z regionami 3"-UTR mRNA (innymi celami), mogą selektywnie i ilościowo (w szczególności podczas usuwania z krążenia produktów genów o niskiej ekspresji) blokować pracę niektórych genów i aktywność innych genów. okazało się, że zestawy profili eksprymowanych mikroRNA (oraz ich cele) zmieniają się dynamicznie podczas ontogenezy, różnicowania komórek i tkanek. Zmiany te są specyficzne zwłaszcza podczas kardiogenezy, procesu optymalizacji wielkości długości dendrytów i liczba synaps komórka nerwowa(w tym miRNA-134, inne małe RNA), rozwój wielu patologii (onkogeneza, niedobory odporności, choroby genetyczne, parkinsonizm, choroba Alzheimera, zaburzenia okulistyczne (siatkówczak itp.) związane z infekcjami o różnym charakterze). Całkowita liczba wykrytych miRNA rośnie znacznie szybciej niż opis ich roli regulacyjnej i powiązania z określonymi celami.
Analiza obliczeniowa przewiduje setki celów mRNA dla poszczególnych miRNA i regulację poszczególnych mRNA przez wiele miRNA. Zatem miRNA mogą służyć do eliminacji transkryptów genów docelowych lub strojenie ich ekspresja na poziomie transkrypcji/translacji. Rozważania teoretyczne i wyniki eksperymentalne potwierdzają istnienie różnorodnych ról miRNA.
Pełniejsza lista aspektów związanych z podstawową rolą małych RNA u eukariontów w procesach wzrostu/rozwoju oraz w niektórych patologiach (w tym w epigenomice nowotworów) znajduje odzwierciedlenie w przeglądzie.
Małe RNA w onkologii
Procesom wzrostu, rozwoju, progresji i przerzutów nowotworów towarzyszy wiele zmian epigenetycznych, które rozwijają się w rzadsze, trwale dziedziczne zmiany genetyczne. Rzadkie mutacje mogą jednak mieć duże znaczenie (dla konkretnego osobnika, nozologia), ponieważ w odniesieniu do poszczególnych genów (np. APC, K-ras, p53) tzw efekt „lejka” związany z niemal nieodwracalnym rozwojem/konsekwencjami nowotworu. Specyficzna dla nowotworu heterogeniczność komórek progenitorowych pod względem profilu ekspresji różnych genów (białek, RNA, małych RNA) jest zdeterminowana przez powiązane różnice w restrukturyzowanych strukturach epigenomicznych. Epigenom modulowany jest poprzez metylację, potranslacyjne modyfikacje/wymianę histonów (na niekanoniczne), przebudowę struktury nukleosomalnej genów/chromatyny (w tym imprinting genomowy, czyli dysfunkcję ekspresji alleli genów rodzicielskich i chromosomów X) ). Wszystko to, przy udziale RNAi regulowanych przez małe RNA, prowadzi do pojawienia się wadliwych struktur heterochromatycznych (w tym hipometylowanych centromerów).
Powstawanie mutacji specyficznych dla genu może być poprzedzone znaną akumulacją setek tysięcy somatycznych mutacji klonalnych w prostych powtórzeniach lub mikrosatelitach regionu niekodującego (rzadko kodującego) - przynajmniej w nowotworach z fenotypem mutatora mikrosatelitarnego (MMP) ; stanowią znaczną część nowotworów jelita grubego, płuc, żołądka, endometrium itp. Niestabilne powtórzenia mikrosatelitarne mono-/heteronukleotydowe (poli-A6-10, podobne) wielokrotnie częściej występują w niekodujących genach regulatorowych, które kontrolują ekspresję (introny, międzygenowe) niż w regionach kodujących (eksonach) genomu nowotworów niestabilnych mikrosatelitarnie, MSI+. Chociaż natura wyglądu i mechanizmy lokalizacji stabilnych/niestabilnych regionów stwardnienia rozsianego nie są do końca jasne, powstawanie niestabilności stwardnienia rozsianego koreluje z częstotliwością mutacji wielu genów, które nie były wcześniej zmutowane w guzach MSI+ i prawdopodobnie kierowały szlakami ich progresja; Co więcej, częstotliwość powtarzających się mutacji MSI w tych nowotworach wzrosła o ponad dwa rzędy wielkości. Nie wszystkie geny analizowano pod kątem obecności powtórzeń, ale stopień ich zmienności w regionach kodujących/niekodujących jest różny, a dokładność metod określania częstotliwości mutacji jest względna. Ważne jest, aby niekodujące regiony powtórzeń zmieniających się w MSI były często bialleliczne, podczas gdy regiony kodujące były monoalleliczne.
Globalny spadek metylacji w nowotworach jest typowy dla powtórzeń, elementów transpozycyjnych (TE; wzrost ich transkrypcji), promotorów, miejsc CpG genów miRNA supresorowych nowotworu i koreluje z hipertranskrypcją retrotranspozonów w postępujących komórkach nowotworowych. Zwykle fluktuacje w „metylomie” są powiązane z „falami metylacji” specyficznymi dla rodzica/etapu/tkanki i silną metylacją centromerowych regionów satelitarnych heterochromatyny, regulowaną przez małe RNA. Gdy satelity są niedometylowane, powstałej niestabilności chromosomów towarzyszy zwiększona rekombinacja, a zakłócenie metylacji ME może wywołać ich ekspresję. Czynniki te sprzyjają rozwojowi fenotypu nowotworu. Terapia małymi RNA może być wysoce specyficzna, ale z tego powodu musi być kontrolowana celem może być nie tylko pojedynczy osobnik, ale także wiele cząsteczek mRNA/RNA i nowo syntetyzowane RNA z różnych (w tym niekodujących powtórzeń międzygenowych) regionów chromosomów.
Większość ludzkiego genomu składa się z powtórzeń i ME. Retrotranspozon L1 (element LINE) zawiera, podobnie jak endogenne retrowirusy, odwrotną (RTazę), endonukleazę i jest potencjalnie zdolny do przenoszenia nieautonomicznych (Alu, SVA itp.) retroelementów; wyciszenie elementów L1/podobnych następuje w wyniku metylacji w miejscach CpG. Należy zauważyć, że wśród miejsc CpG genomu wyspy CpG promotorów genów są słabo zmetylowane, a sama 5-metylocytozyna jest zasadą potencjalnie mutagenną, deaminowaną do tyminy (chemicznie lub przy udziale edycji RNA/(DNA), DNA naprawa); jednakże niektóre wyspy CpG podlegają nadmiernej nieprawidłowej metylacji, której towarzyszy represja genów supresorowych i rozwój raka. Dalej: białko wiążące RNA kodowane przez L1, oddziałując z białkami AGO2 (rodzina Argo-naute) i FMRP (kruche białko upośledzenia umysłowego, białko efektorowego kompleksu RISC), sprzyja ruchowi elementu L1 - co wskazuje na możliwe wzajemna regulacja układów RNAi i retropozycja elementów ludzkiej LINE. Szczególnie ważne jest, aby powtórzenia Alu mogły przemieszczać się do regionu intron/ekson genów.
Te i podobne mechanizmy mogą zwiększać patologiczną plastyczność genomu komórki nowotworowej. Supresji RTazy (kodowanej, podobnie jak endonukleaza, przez elementy L1; RTaza jest również kodowana przez endogenne retrowirusy) poprzez mechanizm RNAi towarzyszyło zmniejszenie proliferacji i zwiększone różnicowanie w wielu liniach komórek nowotworowych. Po wprowadzeniu elementu L1 do protoonkogenu lub genu supresorowego zaobserwowano pęknięcia dwuniciowego DNA. W tkankach linii zarodkowej (myszy/człowiek) poziom ekspresji L1 był podwyższony, a jego metylacja zależała od układu wyciszającego związanego z piRNA (26-30 bp), gdzie białka PIWI są wariantami dużej rodziny białek Argo-naute , mutacje, w których prowadzą do demetylacji/derepresji elementów L1/podobnych z długimi powtórzeniami końcowymi. Białka PIWI w większym stopniu niż białka Dicer-1/2 i Ago są powiązane ze szlakami wyciszania rasiRNA. Szlaki wyciszania, w których pośredniczą piRNA/siRNA, realizowane są poprzez ciałka wewnątrzjądrowe zawierające duże, konserwatywne ewolucyjnie wielobiałkowe kompleksy PcG, których funkcje są często upośledzone w komórkach nowotworowych. Kompleksy te odpowiadają za działanie dalekosiężne (w poprzek ponad 10 kb, pomiędzy chromosomami) i regulują skupisko genów HOX odpowiedzialnych za budowę ciała.
Nowe zasady terapii antysensownej można opracować, biorąc pod uwagę wiedzę na temat bardziej specyficznych (niż modyfikujące histony inhibitory metylacji DNA/białka) przeciwnowotworowych środków epigenomicznych, podstawowe zasady wyciszania epigenomowego RNA i rolę małych RNA w karcynogenezie.
Mikro-RNA w onkologii
Wiadomo, że zwiększonemu wzrostowi nowotworu i przerzutom może towarzyszyć wzrost niektórych i spadek ekspresji innych osobników/zestawów miRNA (Tabela 1). Niektóre z nich mogą odgrywać rolę sprawczą w powstawaniu nowotworów; a nawet te same miRNA (takie jak miR-21/-24) w różnych komórkach nowotworowych mogą wykazywać zarówno właściwości onkogenne, jak i supresyjne. Każdy typ ludzkiego nowotworu złośliwego można wyraźnie odróżnić na podstawie „odcisku palca miRNA”, a niektóre miRNA mogą działać jako onkogeny, supresory nowotworów, inicjatory migracji komórek, inwazji i przerzutów. W tkankach zmienionych patologicznie często stwierdza się zmniejszoną ilość kluczowych miRNA, które prawdopodobnie biorą udział w systemach obrony przeciwnowotworowej. MiRNA (miR) zaangażowane w onkogenezę uformowały ideę tzw. „oncomirah”: analiza ekspresji ponad 200 miRNA w ponad 1000 próbek chłoniaków i nowotworów litych umożliwiła skuteczną klasyfikację nowotworów na podtypy w zależności od ich pochodzenia i stopnia zróżnicowania. Funkcje i rolę miRNA zostały z powodzeniem zbadane przy użyciu: oligonukleotydów anty-miR modyfikowanych (w celu wydłużenia czasu życia) w grupach 2"-O-metylowych i 2"-O-metoksyetylowych; jak również oligonukleotydy LNA, w których atomy tlenu rybozy w pozycjach 2” i 4” są połączone mostkiem metylenowym.
(Tabela 1)……………….
Guz | miRNA |
Rak płuc | 17-92 , niech-7↓ , 124a↓ , 126 ↓ , 143 ↓ , 145 ↓ , 155 , 191 , 205 , 210 |
Rak sutka | 21 , 125b↓ , 145 ↓ , 155 |
Rak prostaty | 15a ↓ , 16-1 ↓ , 21 , 143 ↓ ,145 ↓ |
Rak jelita | 19a , 21 , 143 ↓ , 145 ↓ |
Rak trzustki | 21 , 103 , 107 , 155 w |
Rak jajnika | 210 |
Przewlekła białaczka limfatyczna | 15a ↓ , 16-1 ↓ , 16-2 , 23 B , 24-1 , 29 ↓ , 146 , 155 , 195 , 221 , 223 ↓ |
Tabela 1 .
miRNA, których ekspresja wzrasta () lub maleje ( ↓ ) w niektórych z najczęstszych nowotworów w porównaniu z normalnymi tkankami (patrz i także).
Uważa się, że regulacyjna rola ekspresji, zanikania i amplifikacji genów miRNA w podatności na inicjację, wzrost i progresję większości nowotworów jest znacząca, a mutacje w parach miRNA/docelowy mRNA są zsynchronizowane. Profil ekspresji miRNA można wykorzystać do klasyfikacji, diagnostyki i prognozowania klinicznego w onkologii. Zmiany w ekspresji miRNA mogą wpływać na cykl komórkowy, czyli program przetrwania komórki. Mutacje miRNA w komórkach macierzystych i somatycznych (jak również wybór polimorficznych wariantów celów mRNA) mogą przyczyniać się do wzrostu, progresji i patofizjologii wielu (jeśli nie wszystkich) nowotworów, a nawet odgrywać kluczową rolę we wzroście, postępie i patofizjologii. Za pomocą miRNA możliwa jest korekta apoptozy.
Oprócz pojedynczych miRNA odkryto ich skupiska, działające jako onkogen wywołujący w szczególności rozwój raka tkanki krwiotwórczej u myszy doświadczalnych; Geny miRNA o właściwościach onkogennych i supresorowych mogą znajdować się w tym samym klastrze. Analiza skupień profili ekspresji miRNA w nowotworach pozwala określić jego pochodzenie (nabłonek, tkanka krwiotwórcza itp.) i sklasyfikować różne nowotwory tej samej tkanki o nieidentycznych mechanizmach transformacji. Ocenę profilu ekspresji miRNA można przeprowadzić przy użyciu nano-/mikromacierzy; Dokładność takiej klasyfikacji przy opracowywaniu technologii (co nie jest łatwe) okazuje się większa niż przy zastosowaniu profili mRNA. Niektóre miRNA biorą udział w różnicowaniu komórek krwiotwórczych (myszy, ludzki), inicjowaniu progresji komórek nowotworowych. Ludzkie geny miRNA często zlokalizowane są w tzw. miejsca „kruche”, obszary z przewagą delecji/insercji, przerwy punktowe, translokacje, transpozycje, minimalnie usunięte i amplifikowane regiony heterochromatyny zaangażowane w onkogenezę.
Angiogeneza . Rola miRNA w angiogenezie jest prawdopodobnie znacząca. Zwiększonej angiogenezie w niektórych ludzkich gruczolakorakach aktywowanych Myc towarzyszyły zmiany we wzorze ekspresji niektórych miRNA, a knockdown genów innych miRNA prowadził do osłabienia i zahamowania wzrostu nowotworu. Wzrostowi nowotworu towarzyszyły mutacje w genach K-ras, Myc i TP53, zwiększone wytwarzanie angiogennego czynnika VEGF oraz stopień unaczynienia związanego z Myc; podczas gdy czynniki antyangiogenne Tsp1 i CTGF były tłumione przez miR-17-92 i inne miRNA związane z klastrami. Angiogeneza i unaczynienie nowotworu uległy wzmocnieniu (szczególnie w kolonocytach) poprzez koekspresję dwóch onkogenów, a nie jednego.
Neutralizacja czynnika antyangiogennego LATS2, inhibitora zwierzęcej kinazy cyklinozależnej (CDK2; człowiek/mysz), za pomocą miRNA-372/373 („potencjalne onkogeny”) stymulowała wzrost guza jądra bez uszkadzania genu p53.
Potencjalnymi modulatorami właściwości angiogennych (in vitro/in vivo) są miR-221/222, których celem, receptorami c-Kit (inne), są czynniki angiogenezy śródbłonkowych żylnych komórek HUVEC pępowiny itp. Te miRNA i c-Kit oddziałują na siebie w ramach złożonego cyklu kontrolującego zdolność komórek śródbłonka do tworzenia nowych naczyń włosowatych.
Przewlekła białaczka limfatyczna (CLL). W przewlekłej białaczce limfocytowej B-komórkowej (CLL) obserwuje się obniżony poziom ekspresji genów miR-15a/miR-16-1 (i innych) w regionie 13q14 ludzkiego chromosomu – miejscu najczęstszych nieprawidłowości strukturalnych ( w tym delecje regionu 30 kb), chociaż genom wyrażał setki ludzkich dojrzałych i pre-miRNA. Obydwa miRNA, potencjalnie skuteczne w terapii nowotworów, zawierały antysensowne regiony antyapoptotycznego białka Bcl2, tłumiły jego nadekspresję, stymulowały apoptozę, ale były prawie/całkowicie nieobecne w dwóch trzecich „odbiegających od normy” komórek CLL. Częste mutacje sekwencjonowanych miRNA w komórkach macierzystych/somatycznych zidentyfikowano u 11 z 75 pacjentów (14,7%) z rodzinną predyspozycją do CLL (sposób dziedziczenia nieznany), ale nie u 160 zdrowych pacjentów. Obserwacje te budzą spekulacje na temat bezpośredniego funkcjonowania miRNA w białaczce. Obecnie nie wszystko wiadomo na temat związku między poziomem ekspresji genów miRNA (i ich funkcjami) a innymi genami w komórkach normalnych/nowotworowych.
DokumentZnaczenie. Jedną z nich jest dysfunkcja nerwu twarzowego podczas operacji na śliniance przyusznej obecne problemy i jest określany zarówno na podstawie częstości występowania choroby, jak i znacznej częstotliwości
Dawson Church - geniusz jest w twoich genach medycyna epigenetyczna i nowa biologia intencji książka z biblioteki www e - puzzle ru książka z biblioteki www e - puzzle ru spis treści
KsiążkaEtyka duchowość onkologia hiv p garyaev* podsumowanie enfi
DokumentTen artykuł odzwierciedla Nowy wygląd nad problemem onkologii i zakażenia wirusem HIV w świetle genetyki fal językowych (LWG) i teorii kodowania esencji (ESC) opartej na rosyjskiej i innych realiach społeczno-kulturowych.
Centrum Badań Onkologicznych i Blokhina Odintsova Anastasia Sergeevna nowe schematy chemioterapii w zaawansowanym i nawrotowym raku szyjki macicy 14.01.12 – onkologia
Praca dyplomowa4.4. Oznaczanie genu izoenzymu urydynoglukoronylotransferazy (UGT1A1) w surowicy krwi pacjentek chorych na raka szyjki macicy, które otrzymały chemioterapię pierwszego rzutu z użyciem irynotekanu z pochodnymi platyny 105